Secvență conservată - Conserved sequence

Secvențele sunt aminoacizii pentru resturile 120-180 ale proteinelor. Reziduurile care sunt conservate în toate secvențele sunt evidențiate în gri. Sub fiecare sit (adică poziția) alinierii secvenței de proteine este o cheie care denotă situri conservate (*), site-uri cu înlocuiri conservatoare (:), site-uri cu înlocuiri semi-conservative (.) Și site-uri cu înlocuiri neconservative () .
In biologia evolutionista , secvențe conservate sunt identice sau similare secvențe din acizii nucleici ( ADN și ARN ) sau proteine între specii ( secvențe orthologous ), sau într - un genom ( secvențe paraloage ), sau între donor și receptor taxonomice ( secvențe xenologous ). Conservarea indică faptul că o secvență a fost menținută prin selecție naturală .
O secvență foarte conservată este una care a rămas relativ neschimbată cu mult înapoi în arborele filogenetic și, prin urmare, mult înapoi în timp geologic . Exemple de secvențe foarte conservate includ componentele ARN ale ribozomilor prezente în toate domeniile vieții, secvențele homeobox răspândite printre eucariote și tmRNA din bacterii . Studiul conservării secvențelor se suprapune cu domeniile genomică , proteomică , biologie evolutivă , filogenetică , bioinformatică și matematică .
Istorie
Descoperirea rolului ADN-ului în ereditate și observațiile lui Frederick Sanger privind variația între insulinele animale în 1949, au determinat primii biologi moleculari să studieze taxonomia dintr-o perspectivă moleculară. Studiile din anii 1960 au folosit tehnici de hibridizare a ADN-ului și de reactivitate încrucișată a proteinelor pentru a măsura similitudinea dintre proteinele ortoloage cunoscute , precum hemoglobina și citocromul c . În 1965, Émile Zuckerkandl și Linus Pauling au introdus conceptul de ceas molecular , propunând că ratele constante de înlocuire a aminoacizilor ar putea fi utilizate pentru a estima timpul de când au divergut două organisme . În timp ce filogeniile inițiale se potriveau îndeaproape cu înregistrările fosile , observațiile că unele gene par să evolueze la ritmuri diferite au condus la dezvoltarea teoriilor evoluției moleculare . Comparația Margaret Dayhoff din 1966 a secvențelor de ferodoxină a arătat că selecția naturală ar acționa pentru conservarea și optimizarea secvențelor de proteine esențiale vieții.
Mecanisme
De-a lungul multor generații, secvențele de acid nucleic din genomul unei descendențe evolutive se pot schimba treptat în timp datorită mutațiilor și delețiilor aleatorii . Secvențele pot, de asemenea, să se recombine sau să fie șterse din cauza rearanjărilor cromozomiale . Secvențele conservate sunt secvențe care persistă în genom în ciuda acestor forțe și au rate mai mici de mutație decât rata mutației de fond.
Conservarea se poate produce în codare și non-codare secvențe de acid nucleic. Se crede că secvențele de ADN foarte conservate au valoare funcțională, deși rolul pentru multe secvențe de ADN necodificatoare extrem de conservate este slab înțeles. Măsura în care o secvență este conservată poate fi afectată de presiunile de selecție variate , de robustețea sa la mutație, dimensiunea populației și deriva genetică . Multe secvențe funcționale sunt, de asemenea , modulare , conținând regiuni care pot fi supuse unor presiuni de selecție independente , cum ar fi domeniile de proteine .
Secvența de codificare
În secvențele de codificare, secvența de acid nucleic și aminoacid poate fi conservată în diferite măsuri, deoarece degenerarea codului genetic înseamnă că mutațiile sinonime dintr-o secvență de codificare nu afectează secvența de aminoacizi a produsului său proteic.
Secvențele de aminoacizi pot fi conservate pentru a menține structura sau funcția unei proteine sau a unui domeniu. Proteinele conservate suferă mai puține înlocuiri de aminoacizi sau sunt mai susceptibile de a substitui aminoacizii cu proprietăți biochimice similare . În cadrul unei secvențe, aminoacizii care sunt importanți pentru pliere , stabilitatea structurală sau care formează un situs de legare pot fi mai bine conservați.
Secvența de acid nucleic a unei gene care codifică proteina poate fi, de asemenea, conservată prin alte presiuni selective. Bias utilizării codonului în unele organisme pot limita tipurile de mutații sinonime într - o secvență. Secvențele de acid nucleic care determină structura secundară în ARNm ale unei gene codatoare pot fi selectate împotriva, deoarece unele structuri pot afecta negativ traducerea sau pot fi conservate acolo unde ARNm acționează și ca ARN funcțional necodificator.
Non-codare
Secvențele necodificatoare importante pentru reglarea genelor , cum ar fi siturile de legare sau recunoaștere a ribozomilor și factorii de transcripție , pot fi conservate într-un genom. De exemplu, promotorul unei gene sau operon conservate poate fi, de asemenea, conservat. Ca și în cazul proteinelor, acizii nucleici care sunt importanți pentru structura și funcția ARN necodificator (ncARN) pot fi, de asemenea, conservați. Cu toate acestea, conservarea secvenței în ARNc este în general slabă în comparație cu secvențele de codificare a proteinelor, iar perechile de baze care contribuie la structură sau funcție sunt adesea conservate în schimb.
Identificare
Secvențele conservate sunt de obicei identificate prin abordări bioinformatice bazate pe alinierea secvenței . Progresele în secvențierea ADN de mare viteză și spectrometria de masă a proteinelor au crescut substanțial disponibilitatea secvențelor de proteine și a genomului întreg pentru comparație de la începutul anilor 2000.
Căutare omologică
Secvențele conservați pot fi identificate prin omologie de căutare, folosind instrumente , cum ar fi BLAST , HMMER , OrthologR și Infernal. Instrumentele de căutare a omologiei pot lua ca intrare o secvență individuală de acid nucleic sau proteină sau pot utiliza modele statistice generate din alinieri de secvențe multiple ale secvențelor conexe cunoscute. Modelele statistice, cum ar fi HMM-urile de profil și modelele de covarianță a ARN-ului, care încorporează, de asemenea, informații structurale, pot fi utile atunci când se caută secvențe mai îndepărtate. Secvențele de intrare sunt apoi aliniate cu o bază de date de secvențe de la indivizi înrudiți sau alte specii. Alinierile rezultate sunt apoi punctate pe baza numărului de aminoacizi sau baze potrivite și a numărului de goluri sau ștergeri generate de aliniere. Substituțiile conservatoare acceptabile pot fi identificate folosind matrici de substituție, cum ar fi PAM și BLOSUM . Se presupune că alinierile cu scoruri mari provin din secvențe omoloage. Conservarea unei secvențe poate fi apoi dedusă prin detectarea unor omologi foarte asemănători pe o gamă largă filogenetică.
Alinierea secvenței multiple
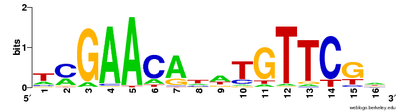
Mai multe alinieri de secvențe pot fi utilizate pentru a vizualiza secvențe conservate. CLUSTAL Formatul include o cheie de text simplu pentru a adnota coloane conservate ale aliniamentului, ceea ce denotă secvența conservată (*), mutații conservatoare (:), mutațiile semi-conservatoare (.), Și mutațiile non-conservatoare () logo - uri secvență poate arăta , de asemenea secvență conservată prin reprezentarea proporțiilor de caractere în fiecare punct al alinierii după înălțime.
Alinierea genomului

Alinierile genomului întreg (WGA) pot fi, de asemenea, utilizate pentru a identifica regiunile extrem de conservate de-a lungul speciilor. În prezent, acuratețea și scalabilitatea instrumentelor WGA rămâne limitată datorită complexității de calcul a gestionării rearanjărilor, regiunilor repetate și dimensiunii mari a multor genomi eucariote. Cu toate acestea, WGA-urile a 30 sau mai multe bacterii strâns legate (procariote) sunt acum din ce în ce mai fezabile.
Sisteme de notare
Alte abordări folosesc măsurători de conservare bazate pe teste statistice care încearcă să identifice secvențe care mută diferit la o rată de mutație de fond (neutră) așteptată.
Cadrul GERP (Genomic Evolutionary Rate Profiling) evaluează conservarea secvențelor genetice între specii. Această abordare estimează rata mutației neutre într-un set de specii dintr-o aliniere de secvență multiplă și apoi identifică regiunile secvenței care prezintă mai puține mutații decât se aștepta. Aceste regiuni sunt apoi atribuite scoruri pe baza diferenței dintre rata de mutație observată și rata de mutație de fond așteptată. Un scor GERP ridicat indică apoi o secvență foarte conservată.
LIST (Local Identity and Shared Taxa) se bazează pe presupunerea că variațiile observate la speciile strâns legate de om sunt mai semnificative la evaluarea conservării în comparație cu cele din speciile înrudite la distanță. Astfel, LIST utilizează identitatea de aliniere locală în jurul fiecărei poziții pentru a identifica secvențe relevante în alinierea de secvențe multiple (MSA) și apoi estimează conservarea pe baza distanțelor de taxonomie ale acestor secvențe față de om. Spre deosebire de alte instrumente, LIST ignoră numărul / frecvența variațiilor din MSA.
Aminode combină mai multe alinieri cu analize filogenetice pentru a analiza modificările proteinelor omoloage și pentru a produce un grafic care indică ratele locale ale modificărilor evolutive. Această abordare identifică regiunile constrânse din punct de vedere evolutiv într-o proteină, care sunt segmente care sunt supuse selecției de purificare și sunt de obicei critice pentru funcția normală a proteinelor.
Alte abordări, cum ar fi PhyloP și PhyloHMM, încorporează metode statistice de filogenetică pentru a compara distribuțiile de probabilitate ale ratelor de substituție, ceea ce permite detectarea atât a conservării, cât și a mutației accelerate. În primul rând, se generează o distribuție a probabilității de fond a numărului de substituții care se așteaptă să aibă loc pentru o coloană într-o aliniere de secvență multiplă, pe baza unui arbore filogenetic . Relațiile evolutive estimate între speciile de interes sunt utilizate pentru a calcula semnificația oricăror substituții (adică o substituție între două specii strâns înrudite poate fi mai puțin probabil să apară decât cele înrudite la distanță și, prin urmare, mai semnificativă). Pentru a detecta conservarea, se calculează o distribuție de probabilitate pentru un subset al alinierii secvenței multiple și se compară cu distribuția de fundal utilizând un test statistic, cum ar fi un test de raport de probabilitate sau un test de scor . Valorile P generate de compararea celor două distribuții sunt apoi utilizate pentru a identifica regiunile conservate. PhyloHMM folosește modele ascunse Markov pentru a genera distribuții de probabilitate. Pachetul software PhyloP compară distribuțiile de probabilitate utilizând un test de raport de probabilitate sau un test de scor , precum și folosind un sistem de notare asemănător GERP.
Conservare extremă
Elemente ultra-conservate
Elementele ultra-conservate sau UCE-urile sunt secvențe care sunt foarte similare sau identice în mai multe grupări taxonomice . Acestea au fost descoperite pentru prima dată la vertebrate și, ulterior, au fost identificate în cadrul taxonilor foarte diferiți. În timp ce originea și funcția UCE sunt slab înțelese, ele au fost folosite pentru a investiga divergențele în timp real în amniote , insecte și între animale și plante .
Gene conservate universal
Cele mai bine conservate gene sunt cele care pot fi găsite în toate organismele. Acestea constau în principal din ARNc și proteine necesare transcripției și traducerii , despre care se presupune că au fost conservate de la ultimul strămoș comun universal al întregii vieți.
Genele sau familiile de gene care s-au dovedit a fi conservate universal includ factori de alungire care leagă GTP , metionină aminopeptidază 2 , serină hidroximetiltransferază și transportori ATP . Componentele mașinilor de transcripție, cum ar fi ARN polimeraza și helicasele , și ale mașinilor de traducere, cum ar fi ARN-urile ribozomale , ARNt -urile și proteinele ribozomale sunt, de asemenea, conservate universal.
Aplicații
Filogenetică și taxonomie
Seturile de secvențe conservate sunt adesea utilizate pentru generarea copacilor filogenetici , deoarece se poate presupune că organismele cu secvențe similare sunt strâns legate. Alegerea secvențelor poate varia în funcție de sfera taxonomică a studiului. De exemplu, cele mai bine conservate gene, cum ar fi ARN-ul 16S și alte secvențe ribozomale, sunt utile pentru reconstituirea relațiilor filogenetice profunde și identificarea filelor bacteriene în studiile de metagenomică . Secvențele care sunt conservate într-o cladă, dar care suferă unele mutații, cum ar fi genele de menaj , pot fi utilizate pentru a studia relațiile dintre specii. Regiunea distanțieră transcrisă intern (ITS), care este necesară pentru distanțarea genelor de ARNr conservate, dar care suferă o evoluție rapidă, este frecvent utilizată pentru a clasifica ciuperci și tulpini de bacterii care evoluează rapid.
Cercetare medicala
Deoarece secvențele foarte conservate au adesea funcții biologice importante, ele pot fi utile ca punct de plecare pentru identificarea cauzei bolilor genetice . Multe tulburări metabolice congenitale și boli de depozitare lizozomale sunt rezultatul modificărilor aduse genelor individuale conservate, rezultând în enzime lipsă sau defecte, care sunt cauza principală a simptomelor bolii. Bolile genetice pot fi prezise prin identificarea secvențelor care sunt conservate între oameni și organismele de laborator, cum ar fi șoarecii sau muștele fructelor , și studierea efectelor eliminării acestor gene. Studiile de asociere la nivel de genom pot fi, de asemenea, utilizate pentru a identifica variațiile în secvențele conservate asociate cu rezultatele bolii sau ale sănătății. În boala Alzehimer au fost descoperite peste două duzini de noi potențiali loci de susceptibilitate
Adnotare funcțională
Identificarea secvențelor conservate poate fi utilizată pentru a descoperi și prezice secvențe funcționale, cum ar fi genele. Secvențele conservate cu o funcție cunoscută, cum ar fi domeniile proteice, pot fi, de asemenea, utilizate pentru a prezice funcția unei secvențe. Bazele de date ale domeniilor de proteine conservate, cum ar fi Pfam și baza de date a domeniului conservat, pot fi utilizate pentru a adnota domenii funcționale în genele de codificare a proteinelor prezise.
Vezi si
- Biologie evolutivă a dezvoltării
- Site separat
- Alinierea secvenței
- Software de aliniere a secvențelor
- UCbase
- Element ultra-conservat
Referințe
