Rosetta @ home - Rosetta@home
 | |
| Dezvoltatori | Laboratorul Baker, Universitatea din Washington; Rosetta Commons |
|---|---|
| Eliberarea inițială | 6 octombrie 2005 |
| Versiune stabila | Rosetta: 4,20 / 1 mai 2020
Rosetta Mini: 3,78 / 3 octombrie 2017 Rosetta pentru Android: 4.20 / 1 mai 2020 |
| Starea de dezvoltare | Activ |
| Sistem de operare | Windows , macOS , Linux , Android |
| Platformă | BOINC |
| Licență | Freeware proprietar pentru utilizare academică și non-profit, licență comercială disponibilă |
| Performanță medie | 487.946 Giga FLOPS |
| Utilizatori activi | 36.726 |
| Total utilizatori | 1.363.584 |
| Gazde active | 249,673 |
| Total gazde | 529.112 |
| Site-ul web | boinc |
Rosetta @ home este un proiect de calcul distribuit pentru predicția structurii proteinelor pe platforma Berkeley Open Infrastructure for Network Computing (BOINC), condusă de laboratorul Baker de la Universitatea din Washington . Rosetta @ home își propune să prezică andocarea proteină-proteină și să proiecteze noi proteine cu ajutorul a aproximativ cincizeci și cinci de mii de calculatoare active voluntare care procesează în medie peste 487.946 Giga FLOPS începând cu 19 septembrie 2020. Foldit , un joc video Rosetta @ home, își propune pentru a atinge aceste obiective cu o abordare crowdsourcing . Deși o mare parte a proiectului este orientată către cercetarea de bază pentru a îmbunătăți acuratețea și robustețea metodelor de proteomică , Rosetta @ home face, de asemenea, cercetări aplicate asupra malariei , bolii Alzheimer și a altor patologii.
Ca toate proiectele BOINC, Rosetta @ home folosește resurse de procesare inactivă de la computerele voluntarilor pentru a efectua calcule pe unități de lucru individuale . Rezultatele finalizate sunt trimise către un server central al proiectului unde sunt validate și asimilate în bazele de date ale proiectului . Proiectul este multiplataforma și rulează pe o mare varietate de configurații hardware. Utilizatorii pot vizualiza progresul predicției individuale a structurii proteinelor pe screensaver-ul Rosetta @ home.
În plus față de cercetările legate de boli, rețeaua Rosetta @ home servește ca cadru de testare pentru noi metode în bioinformatică structurală . Astfel de metode sunt apoi utilizate în alte aplicații bazate pe Rosetta, cum ar fi RosettaDock sau Human Proteome Folding Project și Microbiome Immunity Project , după ce au fost suficient dezvoltate și dovedite stabile pe setul mare și divers de computere voluntare Rosetta @ home. Două teste deosebit de importante pentru noile metode dezvoltate în Rosetta @ home sunt evaluarea critică a tehnicilor de predicție a structurii proteinelor (CASP) și experimentele de evaluare critică a predicției interacțiunilor (CAPRI), experimente bienale care evaluează stadiul tehnicii în structura proteinelor predicție și, respectiv, predicție de andocare proteină-proteină. Rosetta @ home se clasează în mod constant printre cei mai importanți predictori de andocare și este unul dintre cei mai buni predictori de structură terțiară disponibili.
Cu un aflux de noi utilizatori care doresc să participe la lupta împotriva pandemiei COVID-19 , cauzată de SARS-CoV-2 , Rosetta @ home și-a mărit puterea de calcul până la 1,7 PetaFlops începând cu 28 martie 2020. Pe 9 septembrie, În 2020, cercetătorii Rosetta @ home au publicat o lucrare care descrie 10 candidați antivirali puternici împotriva SARS-CoV-2. Rosetta @ home a contribuit la această cercetare și acești candidați antivirali se îndreaptă spre studiile clinice de fază 1, care ar putea începe la începutul anului 2022. Potrivit echipei Rosetta @ home, voluntarii Rosetta au contribuit la dezvoltarea unui vaccin împotriva nanoparticulelor. Acest vaccin a fost autorizat și este cunoscut sub numele de IVX-411 de către Icosavax, care a început un studiu clinic de fază I / II în iunie 2021 și GBP 510, care este dezvoltat de SK Bioscience și este deja aprobat pentru un studiu clinic de fază III în South Coreea.
NL-201 , un candidat la cancer care a fost creat pentru prima dată la Institutul de Proiectare a Proteinelor (IPD) și publicat într-o lucrare din ianuarie 2019, a început un studiu clinic de fază 1 uman în mai 2021 cu sprijinul Neoleukin Therapeutics, el însuși un spin- oprit din IPD. Rosetta @ home a jucat un rol în dezvoltarea NL-201 și a contribuit cu experimente de „pliere înainte” care au ajutat la validarea modelelor de proteine.
Platforma de calcul
Aplicația Rosetta @ home și platforma de calcul distribuită BOINC sunt disponibile pentru sistemele de operare Windows , Linux și macOS ; BOINC rulează și pe mai multe altele, de exemplu, FreeBSD. Participarea la Rosetta @ home necesită o unitate centrală de procesare (CPU) cu o viteză de ceas de cel puțin 500 MHz , 200 megabiți de spațiu liber pe disc , 512 megabiți de memorie fizică și conectivitate la internet. Începând cu 20 iulie 2016, versiunea actuală a aplicației Rosetta Mini este 3.73. Versiunea actuală recomandată a programului BOINC este 7.6.22. Protocolul standard de transfer hipertext (HTTP) ( portul 80) este utilizat pentru comunicarea între clientul BOINC al utilizatorului și serverele Rosetta @ home de la Universitatea din Washington; HTTPS (portul 443) este utilizat în timpul schimbului de parole. Controlul la distanță și local al clientului BOINC utilizează portul 31416 și portul 1043, care ar putea fi necesar să fie deblocate în mod specific dacă se află în spatele unui firewall . Unitățile de lucru care conțin date despre proteine individuale sunt distribuite de pe servere situate în laboratorul Baker de la Universitatea din Washington către computerele voluntarilor, care apoi calculează o predicție de structură pentru proteina atribuită. Pentru a evita predicțiile de structură duplicate pe o anumită proteină, fiecare unitate de lucru este inițializată cu un număr de semințe aleatorii . Acest lucru oferă fiecărei predicții o traiectorie unică de coborâre de-a lungul peisajului energetic al proteinei . Predicțiile privind structura proteinelor din Rosetta @ home sunt aproximări ale unui minim global în peisajul energetic al unei proteine date. Acest minim global reprezintă cea mai favorabilă conformație energetică a proteinei, adică starea sa nativă .
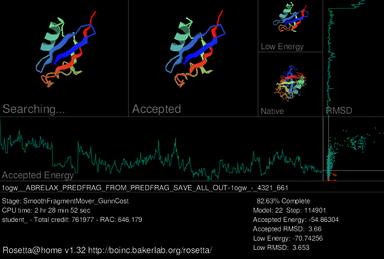
O caracteristică principală a interfeței grafice de utilizator (GUI) Rosetta @ home este un screensaver care arată progresul unei unități de lucru curente în timpul procesului de pliere a proteinelor simulate . În stânga sus a screensaver-ului curent, proteina țintă este prezentată adoptând diferite forme (conformații) în căutarea celei mai scăzute structuri energetice. Reprezentată imediat în dreapta este structura celui mai recent acceptat. În partea dreaptă sus este afișată cea mai mică conformație energetică a momelului curent; sub aceasta se află structura adevărată sau nativă a proteinei, dacă a fost deja determinată. Trei grafice sunt incluse în screensaver. Aproape de mijloc, este afișat un grafic pentru energia liberă termodinamică a modelului acceptat , care fluctuează pe măsură ce modelul acceptat se schimbă. Un grafic al deviației rădăcină-medie-pătrat (RMSD) a modelului acceptat, care măsoară cât de similar este structural modelul acceptat cu modelul nativ, este prezentat în extrema dreaptă. În partea dreaptă a graficului de energie acceptat și sub graficul RMSD, rezultatele acestor două funcții sunt utilizate pentru a produce o diagramă de energie vs. RMSD, deoarece modelul este rafinat progresiv.
Ca toate proiectele BOINC, Rosetta @ home rulează în fundalul computerului utilizatorului, utilizând alimentarea computerului inactiv, fie la, fie înainte de a se conecta la un cont pe sistemul de operare gazdă . Programul eliberează resurse de la CPU, deoarece acestea sunt necesare pentru alte aplicații, astfel încât utilizarea normală a computerului să nu fie afectată. Multe setări ale programului pot fi specificate prin intermediul preferințelor contului utilizatorului, inclusiv: procentul maxim de resurse CPU pe care programul le poate utiliza (pentru a controla consumul de energie sau producția de căldură de pe un computer care rulează la o capacitate susținută), orele zilei în care programul poate rula , si multe altele.
Rosetta, software-ul care rulează pe rețeaua Rosetta @ home, a fost rescris în C ++ pentru a permite o dezvoltare mai ușoară decât cea permisă de versiunea sa originală, care a fost scrisă în Fortran . Această nouă versiune este orientată obiect și a fost lansată pe 8 februarie 2008. Dezvoltarea codului Rosetta este realizată de Rosetta Commons. Software-ul este licențiat gratuit comunității academice și disponibil companiilor farmaceutice contra cost.
Semnificația proiectului
Odată cu proliferarea proiectelor de secvențiere a genomului , oamenii de știință pot deduce secvența de aminoacizi sau structura primară a multor proteine care îndeplinesc funcții în interiorul celulei. Pentru a înțelege mai bine funcția unei proteine și pentru a ajuta la proiectarea rațională a medicamentelor , oamenii de știință trebuie să cunoască structura terțiară tridimensională a proteinei .
Structurile 3D ale proteinelor sunt determinate în mod experimental prin cristalografie cu raze X sau spectroscopie cu rezonanță magnetică nucleară (RMN). Procesul este lent (poate dura săptămâni sau chiar luni pentru a afla cum să cristalizeze o proteină pentru prima dată) și costisitor (în jur de 100.000 USD pe proteină). Din păcate, rata la care sunt descoperite noi secvențe depășește cu mult rata de determinare a structurii - din mai mult de 7.400.000 de secvențe de proteine disponibile în baza de date proteică nonredundantă (NCBI) a Centrului Național pentru Informații despre Biotehnologie (NCBI), mai puțin de 52.000 de structuri 3D de proteine au fost rezolvate și depozitate în Protein Data Bank , principalul depozit pentru informații structurale despre proteine. Unul dintre obiectivele principale ale Rosetta @ home este de a prezice structurile proteice cu aceeași acuratețe ca metodele existente, dar într-un mod care necesită mult mai puțin timp și bani. Rosetta @ home dezvoltă, de asemenea, metode pentru a determina structura și andocarea proteinelor de membrană (de exemplu, receptorii cuplați cu proteina G (GPCR)), care sunt extrem de dificil de analizat cu tehnici tradiționale precum cristalografia cu raze X și spectroscopia RMN, dar reprezintă majoritatea de ținte pentru drogurile moderne.
Progresul în predicția structurii proteinelor este evaluat în experimentul bianual de evaluare critică a tehnicilor de predicție a structurii proteinelor (CASP), în care cercetătorii din întreaga lume încearcă să obțină structura unei proteine din secvența de aminoacizi a proteinei. Grupurile cu punctaje mari din acest experiment uneori competitiv sunt considerate de facto purtători de standarde pentru ceea ce este stadiul tehnicii în predicția structurii proteinelor. Rosetta, programul pe care se bazează Rosetta @ home, a fost utilizat de la CASP5 în 2002. În experimentul CASP6 din 2004, Rosetta a făcut istorie fiind primul care a produs o rezoluție apropiată de nivelul atomic, predicția structurii proteinei ab initio în prezentat model pentru ținta CASP T0281. Modelarea ab initio este considerată o categorie deosebit de dificilă de predicție a structurii proteinelor, deoarece nu folosește informații din omologia structurală și trebuie să se bazeze pe informațiile din omologia secvenței și modelarea interacțiunilor fizice în cadrul proteinei. Rosetta @ home a fost utilizat în CASP din 2006, unde a fost printre predictorii de top din fiecare categorie de predicție a structurii în CASP7. Aceste predicții de înaltă calitate au fost permise de puterea de calcul pusă la dispoziție de voluntarii Rosetta @ home. Creșterea puterii de calcul permite Rosetta @ home să probeze mai multe regiuni de spațiu de conformație (posibilele forme pe care le poate asuma o proteină), care, potrivit paradoxului lui Levinthal , se prezice că va crește exponențial odată cu lungimea proteinei.
Rosetta @ home este, de asemenea, utilizat în predicția de andocare proteină-proteină , care determină structura mai multor proteine complexate sau structura cuaternară . Acest tip de interacțiune proteică afectează multe funcții celulare, inclusiv legarea antigen-anticorp și enzimă-inhibitor și importul și exportul celular. Determinarea acestor interacțiuni este esențială pentru proiectarea medicamentelor . Rosetta este utilizată în experimentul Evaluare critică a predicției interacțiunilor (CAPRI), care evaluează starea câmpului de andocare a proteinelor similar cu modul în care măsurătorile CASP progresează în predicția structurii proteinelor. Puterea de calcul pusă la dispoziție de voluntarii proiectului Rosetta @ home a fost citată ca un factor major în performanța Rosetta în CAPRI, unde predicțiile sale de andocare au fost printre cele mai exacte și complete.
La începutul anului 2008, Rosetta a fost utilizată pentru a proiecta prin calcul o proteină cu o funcție care nu a fost observată până acum în natură. Acest lucru a fost inspirat parțial de retragerea unei hârtii cu profil înalt din 2004, care a descris inițial proiectarea de calcul a unei proteine cu activitate enzimatică îmbunătățită în raport cu forma sa naturală. Lucrarea de cercetare din 2008 a grupului David Baker care descrie modul în care a fost fabricată proteina, care a citat Rosetta @ home pentru resursele de calcul pe care le-a pus la dispoziție, a reprezentat o dovadă importantă a conceptului pentru această metodă de proiectare a proteinelor. Acest tip de proiectare a proteinelor ar putea avea aplicații viitoare în descoperirea medicamentelor, chimia verde și bioremediere .
În plus față de cercetarea de bază în prezicerea structurii, andocării și proiectării proteinelor, Rosetta @ home este, de asemenea, utilizat în cercetarea imediată a bolilor. Numeroase proiecte minore de cercetare sunt descrise în jurnalul David Baker Rosetta @ home. Începând cu februarie 2014, informațiile despre publicațiile recente și o scurtă descriere a lucrării sunt actualizate pe forum. Firul forumului nu mai este utilizat din 2016, iar știrile despre cercetare pot fi găsite în secțiunea de știri generale a proiectului.
Boala Alzheimer
O componentă a suitei software Rosetta, RosettaDesign, a fost utilizată pentru a prezice cu precizie ce regiuni ale proteinelor amiloidogene au cel mai probabil să producă fibrile asemănătoare amiloidului . Luând hexapeptide (șase fragmente lungi de aminoacizi) ale unei proteine de interes și selectând cea mai mică energie potrivită cu o structură similară cu cea a unei hexapeptide care formează fibrile cunoscute, RosettaDesign a reușit să identifice peptidele de două ori mai susceptibile de a forma fibrile ca aleatorii. proteine. Rosetta @ home a fost utilizat în același studiu pentru a prezice structurile beta amiloid , o proteină care formează fibrile care a fost postulată pentru a provoca boala Alzheimer . Rezultate preliminare, dar încă nepublicate, au fost produse pe proteine proiectate de Rosetta, care pot preveni formarea fibrilelor, deși nu se știe dacă poate preveni boala.
Antrax
O altă componentă a Rosetta, RosettaDock, a fost utilizată împreună cu metode experimentale pentru a modela interacțiunile dintre trei proteine - factor letal (LF), factor de edem (EF) și antigen de protecție (PA) - care alcătuiesc toxina antrax . Modelul computerului a prezis cu acuratețe andocarea între LF și PA, ajutând la stabilirea domeniilor proteinelor respective implicate în complexul LF-PA. Această perspectivă a fost utilizată în cele din urmă în cercetări care au dus la îmbunătățirea vaccinurilor antrax.
Virusul herpes simplex 1
RosettaDock a fost utilizat pentru modelarea andocării între un anticorp ( imunoglobulină G ) și o proteină de suprafață exprimată de virusul de la nivelul herpesului , virusul herpes simplex 1 (HSV-1) care servește la degradarea anticorpului antiviral. Complexul proteic prezis de RosettaDock a fost îndeaproape de acord cu modelele experimentale deosebit de dificil de obținut, conducând cercetătorii la concluzia că metoda de andocare are potențialul de a aborda unele dintre problemele pe care cristalografia cu raze X le are cu modelarea interfețelor proteină-proteină.
HIV
Ca parte a cercetării finanțate de un grant de 19,4 milioane de dolari de la Fundația Bill & Melinda Gates , Rosetta @ home a fost utilizată în proiectarea mai multor vaccinuri posibile pentru virusul imunodeficienței umane ( HIV ).
Malarie
În cercetările implicate în inițiativa „ Marile provocări în sănătatea globală” , Rosetta a fost utilizată pentru a proiecta pe bază de calcul noi proteine de endonuclează homing , care ar putea eradica Anopheles gambiae sau ar putea face ca țânțarul să nu poată transmite malaria . Posibilitatea de a modela și modifica interacțiunile proteină-ADN în mod specific, precum cele ale endonucleazelor homing, conferă metodelor de proiectare a proteinelor de calcul precum Rosetta un rol important în terapia genică (care include posibile tratamente pentru cancer).
COVID-19
Suita de modelare moleculară Rosetta a fost recent utilizată pentru a prezice cu precizie structura la scară atomică a proteinei vârf SARS-CoV-2 cu săptămâni înainte de a putea fi măsurată în laborator. Pe 26 iunie 2020, proiectul a anunțat că a reușit să creeze proteine antivirale care neutralizează virionii SARS-CoV-2 în laborator și că aceste medicamente antivirale experimentale sunt optimizate pentru testele pe animale.
Într-o continuare, o lucrare care descrie 10 inhibitori ai miniproteinei SARS-CoV-2 a fost publicată în Science pe 9 septembrie. Doi dintre acești inhibitori, LCB1 și LCB3, sunt de câteva ori mai puternici decât cei mai buni anticorpi monoclonali care sunt dezvoltați împotriva SARS-CoV -2, atât pe bază molară, cât și pe bază de masă. În plus, cercetarea sugerează că acești inhibitori își păstrează activitatea la temperaturi ridicate, sunt de 20 de ori mai mici decât un anticorp și, prin urmare, au de 20 de ori mai multe situri potențiale de neutralizare, crescând eficacitatea potențială a unui medicament administrat local. Se așteaptă ca dimensiunea redusă și stabilitatea ridicată a inhibitorilor să le facă adecvate unei formulări de gel care poate fi aplicată nazal sau sub formă de pulbere care trebuie administrată direct pe sistemul respirator. Cercetătorii vor lucra la dezvoltarea acestor inhibitori în terapeutică și profilactică în lunile următoare. Începând cu iulie 2021, se preconizează că acești candidați antivirali vor începe studiile clinice la începutul anului 2022 și primiseră finanțare de la Fundația Bill & Melinda Gates pentru studii clinice preclinice și timpurii. În studiile de testare pe animale, acești candidați antivirali au fost eficienți împotriva variantelor de îngrijorare, inclusiv Alpha, Beta și Gamma.
Rosetta @ home a fost folosit pentru a ajuta la screening-ul celor peste 2 milioane de proteine care leagă SARS-CoV-2 Spike, care au fost proiectate pe bază de calcul și, prin urmare, au contribuit la această cercetare.
Conform echipei Rosetta @ home de la Institutul de Proiectare a Proteinelor, voluntarii Rosetta @ home au contribuit la dezvoltarea candidaților la medicamente antivirale și la un vaccin cu nanoparticule proteice. Vaccinul IVX-411 se află deja într-un studiu clinic de fază 1 condus de Icosavax, în timp ce același vaccin, licențiat către un alt producător și sub numele GBP510, a fost aprobat în Coreea de Sud pentru un studiu de fază III derulat de SK Bioscience . Antiviralele candidate se îndreaptă, de asemenea, spre studiile clinice de fază 1.
Cancer
Cercetătorii Rosetta @ home au proiectat un agonist al receptorului IL-2 numit Neoleukin-2/15 care nu interacționează cu subunitatea alfa a receptorului. Astfel de molecule semnal de imunitate sunt utile în tratamentul cancerului. În timp ce IL-2 natural suferă de toxicitate datorită interacțiunii cu subunitatea alfa, proteina proiectată este mult mai sigură, cel puțin la modelele animale. Rosetta @ home a contribuit la „experimente de pliere înainte” care au ajutat la validarea proiectelor.
Într-o caracteristică din septembrie 2020 în New Yorker , David Baker a declarat că Neoleukin-2/15 va începe studiile clinice la om "mai târziu în acest an". Neoleukin-2/15 este dezvoltat de Neoleukin , o companie derivată din laboratorul Baker. În decembrie 2020, Neoleukin a anunțat că va depune o cerere de investigație pentru medicamente noi la Food and Drug Administration pentru a începe un studiu clinic de fază 1 cu NL-201, care este o dezvoltare ulterioară a Neoleukin-2/15. O cerere similară a fost depusă în Australia și Neoleukin speră să înscrie 120 de participanți la studiul clinic de fază 1. Studiul clinic de fază 1 la om a început pe 5 mai 2021.
Istoria și ramurile dezvoltării
Introdusă inițial de laboratorul Baker în 1998 ca o abordare ab initio a predicției structurii, Rosetta s-a ramificat de atunci în mai multe fluxuri de dezvoltare și servicii distincte. Platforma Rosetta își trage numele de la piatra Rosetta , deoarece încearcă să descifreze „semnificația” structurală a secvențelor de aminoacizi ale proteinelor. La mai bine de șapte ani de la prima apariție a Rosettei, proiectul Rosetta @ home a fost lansat (adică, anunțat că nu mai este beta ) pe 6 octombrie 2005. Mulți dintre studenții absolvenți și alți cercetători implicați în dezvoltarea inițială a Rosettei s-au mutat de atunci în alte universități și instituții de cercetare și, ulterior, au îmbunătățit diferite părți ale proiectului Rosetta.
RosettaDesign
RosettaDesign, o abordare de calcul pentru proiectarea de proteine pe baza Rosetta, a început în 2000 , cu un studiu în reproiectare calea de pliere a proteinei G . In 2002 RosettaDesign a fost utilizat pentru a proiecta Top7 , un 93-aminoacizi lung α / β proteină care a avut un ansamblu ori niciodată înainte înregistrată în natură. Această nouă conformație a fost prezisă de Rosetta la 1,2 Å RMSD de structura determinată de cristalografia cu raze X , reprezentând o predicție neobișnuit de precisă a structurii. Rosetta și RosettaDesign au câștigat o recunoaștere pe scară largă, fiind primii care proiectează și prezic cu exactitate structura unei proteine noi de o asemenea lungime, după cum se reflectă în lucrarea din 2002 care descrie abordarea duală care determină două litere pozitive în revista Science și fiind citată de mai mult de Alte 240 de articole științifice. Produsul vizibil al acestei cercetări, Top7 , a fost prezentat ca „Molecula lunii” a RCSB PDB în octombrie 2006; o superpunere a nucleelor respective (reziduurile 60-79) ale structurilor sale de cristal prevazute și cu raze X sunt prezentate în sigla Rosetta @ home.
Brian Kuhlman, fost asociat postdoctoral în laboratorul lui David Baker și acum profesor asociat la Universitatea din Carolina de Nord, Chapel Hill , oferă RosettaDesign ca serviciu online.
RosettaDock
RosettaDock a fost adăugat la suita de software Rosetta în timpul primului experiment CAPRI din 2002, ca algoritm al laboratorului Baker pentru predicția de andocare proteină-proteină . În acel experiment, RosettaDock a făcut o predicție de înaltă precizie pentru andocarea dintre exotoxina piogenă streptococică A și un lanț β al receptorilor celulelor T și o predicție de precizie medie pentru un complex între α-amilaza porcină și un anticorp camelid . În timp ce metoda RosettaDock a făcut doar două predicții acceptabile și precise din șapte posibile, acest lucru a fost suficient pentru a o clasa pe locul șapte din nouăsprezece metode de predicție în prima evaluare CAPRI.
Dezvoltarea RosettaDock a divizat în două ramuri pentru rundele CAPRI ulterioare, în timp ce Jeffrey Gray, care a pus bazele RosettaDock în timp ce era la Universitatea din Washington , a continuat să lucreze la metodă în noua sa poziție la Universitatea Johns Hopkins . Membrii laboratorului Baker au dezvoltat în continuare RosettaDock în absența lui Gray. Cele două versiuni au diferit ușor în ceea ce privește modelarea lanțului lateral, selecția momeală și alte domenii. În ciuda acestor diferențe, atât metodele Baker, cât și metodele Grey au avut rezultate bune în cea de-a doua evaluare CAPRI, plasând al cincilea și respectiv al șaptelea din 30 de grupuri de predictori. Serverul RosettaDock al lui Jeffrey Gray este disponibil ca serviciu gratuit de predicție de andocare pentru utilizare necomercială.
În octombrie 2006, RosettaDock a fost integrat în Rosetta @ home. Metoda a folosit o fază de modelare de andocare rapidă, brută, folosind doar coloana vertebrală proteică . Aceasta a fost urmată de o fază lentă de rafinare a atomului complet, în care orientarea celor două proteine care interacționează una față de cealaltă și interacțiunile lanțului lateral la interfața proteină-proteină, au fost simultan optimizate pentru a găsi cea mai scăzută conformație energetică. Puterea de calcul mult crescută oferită de rețeaua Rosetta @ home, combinată cu reprezentări revizuite cu arborele de pliere pentru flexibilitatea coloanei vertebrale și modelarea buclelor , au făcut din RosettaDock șasea din 63 de grupuri de predicție în a treia evaluare CAPRI.
Robetta
Serverul Robetta (Rosetta Beta) este un serviciu automat de predicție a structurii proteinelor oferit de laboratorul Baker pentru ab initio necomercial și modelare comparativă. A participat ca server automat de predicție la experimentele CASP bianuale de la CASP5 în 2002, performând printre cele mai bune din categoria de predicție a serverului automat. De atunci, Robetta a concurat în CASP6 și 7, unde s-a descurcat mai bine decât media atât în rândul serverelor automatizate, cât și al grupurilor de predictori umani. De asemenea, participă la evaluarea continuă CAMEO3D .
În modelarea structurii proteinelor începând cu CASP6, Robetta caută mai întâi omologii structurali folosind BLAST , PSI-BLAST și 3D-Jury , apoi analizează secvența țintă în domeniile sale individuale sau îndoirea independentă a unităților de proteine, prin potrivirea secvenței cu familiile structurale. în baza de date Pfam . Domeniile cu omologi structurali urmează apoi un protocol „model bazat pe șabloane” (adică, modelare omologică ). Aici, programul de aliniere intern al laboratorului Baker, K * sync, produce un grup de omologi de secvență și fiecare dintre acestea este modelat prin metoda Rosetta de novo pentru a produce un momeal (posibilă structură). Predicția structurii finale este selectată luând cel mai mic model de energie , determinat de o funcție de energie Rosetta cu rezoluție mică. Pentru domeniile care nu au detectat omologi structurali, este urmat un protocol de novo în care modelul cu cea mai mică energie dintr-un set de momeli generate este selectat ca predicție finală. Aceste predicții de domeniu sunt apoi conectate împreună pentru a investiga interacțiunile inter-domenii, la nivel terțiar, în cadrul proteinei. În cele din urmă, contribuțiile lanțului lateral sunt modelate folosind un protocol pentru căutarea conformațională Monte Carlo .
În CASP8, Robetta a fost mărită pentru a utiliza metoda de rafinare completă a atomilor de înaltă rezoluție a lui Rosetta, absența căreia a fost citată ca principală cauză pentru Robetta fiind mai puțin precisă decât rețeaua Rosetta @ home din CASP7. În CASP11, a fost adăugată o modalitate de a prezice harta de contact a proteinelor prin co-evoluția reziduurilor din proteinele înrudite numită GREMLIN, permițând mai multe succese de novo de ori.
Împăturește-l
La 9 mai 2008, după ce utilizatorii Rosetta @ home au sugerat o versiune interactivă a programului de calcul distribuit , laboratorul Baker a lansat public Foldit , un joc online de predicție a structurii proteinelor bazat pe platforma Rosetta. Începând cu 25 septembrie 2008, Foldit avea peste 59.000 de utilizatori înregistrați. Jocul oferă utilizatorilor un set de controale (de exemplu, se agită, se mișcă, se reconstruiește) pentru a manipula lanțurile vertebrale și lanțurile laterale de aminoacizi ale proteinei țintă în conformații mai favorabile energetic. Utilizatorii pot lucra la soluții individual ca soliști sau colectiv ca evolutori , acumulând puncte în ambele categorii pe măsură ce își îmbunătățesc predicțiile de structură.
Comparație cu proiecte de calcul distribuite similare
Există mai multe proiecte computerizate distribuite care au zone de studiu similare cu cele ale Rosetta @ home, dar diferă în abordarea lor de cercetare:
Pliant @ acasă
Dintre toate proiectele de calcul distribuite majore implicate în cercetarea proteinelor, Folding @ home este singurul care nu folosește platforma BOINC . Atât Rosetta @ home, cât și Folding @ home studiază bolile care se pliază greșit, cum ar fi boala Alzheimer , dar Folding @ home face mult mai exclusiv. Folding @ home folosește aproape în mod exclusiv modele de dinamică moleculară atomică pentru a înțelege cum și de ce proteinele se pliază (sau pot fi greșite și, ulterior, se agregă pentru a provoca boli). Cu alte cuvinte, puterea Folding @ home modelează procesul de pliere a proteinelor, în timp ce puterea Rosetta @ home calculează proiectarea proteinelor și prezice structura și andocarea proteinelor.
Unele dintre rezultatele Rosetta @ home sunt utilizate ca bază pentru unele proiecte Folding @ home. Rosetta oferă cea mai probabilă structură, dar nu este clar dacă aceasta este forma pe care o ia molecula sau dacă este viabilă sau nu. Folding @ home poate fi apoi utilizat pentru a verifica rezultatele Rosetta @ home și poate oferi informații suplimentare la nivel atomic și detalii despre modul în care molecula își schimbă forma.
De asemenea, cele două proiecte diferă semnificativ în ceea ce privește puterea de calcul și diversitatea gazdei. În medie de 6.650 tera FLOPS dintr-o bază gazdă de unități centrale de procesare (procesoare), unități de procesare grafică (GPU-uri) și (anterior) PS3-uri , Folding @ home are de aproape 108 ori mai multă putere de calcul decât Rosetta @ home.
Grila comunitară mondială
Atât faza I, cât și faza II a Proiectului de pliere a proteinelor umane (HPF), un subproiect al World Community Grid , au folosit programul Rosetta pentru a face adnotări structurale și funcționale ale diferitelor genomi . Deși acum îl folosește pentru a crea baze de date pentru biologi, Richard Bonneau , om de știință al Human Proteome Folding Project, a fost activ în dezvoltarea inițială a Rosettei la laboratorul lui David Baker în timp ce obținea doctoratul. Mai multe informații despre relația dintre HPF1, HPF2 și Rosetta @ home pot fi găsite pe site-ul Richard Bonneau.
Predictor @ home
La fel ca Rosetta @ home, Predictor @ home s-a specializat în predicția structurii proteinelor. În timp ce Rosetta @ home folosește programul Rosetta pentru predicția structurii sale, Predictor @ home a folosit metodologia dTASSER. În 2009, Predictor @ home a fost închis.
Alte proiecte de calcul distribuite legate de proteine pe BOINC includ QMC @ home , Docking @ home , POEM @ home , SIMAP și TANPAKU . RALPH @ home, proiectul alfa Rosetta @ home care testează versiuni noi de aplicații, unități de lucru și actualizări înainte de a trece la Rosetta @ home, rulează și pe BOINC.
Contribuții voluntare
Rosetta @ home depinde de puterea de calcul donată de membrii individuali ai proiectului pentru cercetarea sa. Începând cu 28 martie 2020, aproximativ 53.000 de utilizatori din 150 de țări erau membri activi ai Rosetta @ home, contribuind împreună la timpul procesorului inactiv de la aproximativ 54.800 de computere pentru o performanță medie combinată de peste 1,7 Peta FLOPS .

Utilizatorilor li se acordă credite BOINC ca măsură a contribuției lor. Creditul acordat pentru fiecare unitate de lucru este numărul de momeli produse pentru acea unitate de lucru înmulțit cu creditul mediu revendicat pentru momealele prezentate de toate gazdele computerului pentru acea unitate de lucru. Acest sistem personalizat a fost conceput pentru a aborda diferențele semnificative între creditul acordat utilizatorilor cu clientul BOINC standard și un client BOINC optimizat și diferențele de credit între utilizatorii care rulează Rosetta @ home pe sistemele de operare Windows și Linux . Valoarea creditului acordat pe secundă de lucru cu CPU este mai mică pentru Rosetta @ home decât majoritatea celorlalte proiecte BOINC. Rosetta @ home este al treisprezecelea din cele peste 40 de proiecte BOINC în ceea ce privește creditul total.
Utilizatorii Rosetta @ home care prezic structuri proteice prezentate pentru experimentul CASP sunt recunoscuți în publicațiile științifice cu privire la rezultatele lor. Utilizatorii care prezic cea mai mică structură energetică pentru o anumită unitate de lucru sunt prezentați pe pagina de pornire Rosetta @ ca Predictor al zilei , împreună cu orice echipă din care fac parte. Un utilizator al zilei este ales în mod aleatoriu în fiecare zi pentru a fi și pe pagina de pornire, dintre utilizatorii care au creat un profil Rosetta @ home.
Referințe
linkuri externe
- Site oficial
- Baker Lab site - ul Baker Lab
- Jurnalul lui David Baker Rosetta @ home
- BOINC Include o prezentare generală a platformei și un ghid pentru instalarea BOINC și atașarea la Rosetta @ home
- BOINCstats - Rosetta @ home Statistici detaliate despre contribuții
- RALPH @ home Site web pentru proiectul de testare alfa Rosetta @ home
- Videoclip Rosetta @ home pe YouTube Prezentare generală a Rosetta @ home oferit de David Baker și membrii laboratorului
- Rosetta Commons Academic colaborativ pentru dezvoltarea platformei Rosetta
- Pagina web a laboratorului Kuhlman , casa RosettaDesign
Servicii Rosetta online
- Lista Rosetta Commons a serverelor disponibile
- Server de predicție a structurii proteinei Robetta
- ROSIE Docking, proiectare, etc. set de servere multifuncțional
- Server de proiectare RosettaDesign Protein
- RosettaBackrub Server de proiectare a coloanei vertebrale flexibile / proteine


