Gena esențială - Essential gene
Gene esentiale sunt indispensabile gene pentru organisme să crească și să reproduceți urmași sub anumit mediu. Cu toate acestea, a fi esențial depinde în mare măsură de circumstanțele în care trăiește un organism. De exemplu, o genă necesară pentru a digera amidonul este esențială numai dacă amidonul este singura sursă de energie. Recent, s-au făcut încercări sistematice de identificare a acelor gene care sunt absolut necesare pentru a menține viața, cu condiția ca toți nutrienții să fie disponibili. Astfel de experimente au condus la concluzia că numărul absolut necesar de gene pentru bacterii este de ordinul a aproximativ 250-300. Genele esențiale ale organismelor unicelulare codifică proteinele pentru trei funcții de bază, inclusiv prelucrarea informațiilor genetice, plicurile celulare și producerea de energie. Aceste funcții genetice sunt utilizate pentru a menține un metabolism central , pentru a reproduce ADN-ul , pentru a traduce genele în proteine , pentru a menține o structură celulară de bază și pentru a media procesele de transport în și din celulă. Comparativ cu organismele unicelulare, organismele multicelulare au gene mai esențiale legate de comunicare și dezvoltare. Majoritatea genelor esențiale din virusuri sunt legate de prelucrarea și menținerea informațiilor genetice. Spre deosebire de majoritatea organismelor unicelulare, virușilor le lipsesc multe gene esențiale pentru metabolism, ceea ce îi obligă să deturneze metabolismul gazdei. Majoritatea genelor nu sunt esențiale, dar transmit avantaje selective și condiții fizice sporite . Prin urmare, marea majoritate a genelor nu sunt esențiale și multe pot fi șterse fără consecințe, cel puțin în majoritatea circumstanțelor.
Bacterii: studii la nivel de genom
Au fost folosite două strategii principale pentru identificarea genelor esențiale la nivel de genom: ștergerea direcționată a genelor și mutageneza aleatorie utilizând transpozoni . În primul caz, genele individuale adnotate (sau ORF ) sunt complet șterse din genom într-un mod sistematic. În mutageneza mediată de transpozoni, transpozonii sunt introduși aleatoriu în cât mai multe poziții într-un genom posibil, cu scopul de a perturba funcția genelor vizate (a se vedea figura de mai jos). Mutanții de inserție care sunt încă capabili să supraviețuiască sau să crească sugerează transpozonul inserat într-o genă care nu este esențială pentru supraviețuire. Localizarea inserțiilor transposonice poate fi determinată prin hibridizare la microarrays sau prin secvențierea transposonului . Un rezumat al acestor ecrane este prezentat în tabel.
| Organism | Mutageneză | Metodă | Citi cu voce tare | ORF-uri | Non-ess. | Esenţial | % Ess. | Note | Ref. |
|---|---|---|---|---|---|---|---|---|---|
| Mycoplasma genitalium / pneumoniae | Aleatoriu | Populația | Secvențierea | 482 | 130 | 265–350 | 55–73% | --- | |
| Mycoplasma genitalium | Aleatoriu | Clone | Secvențierea | 482 | 100 | 382 | 79% | b, c | |
| Staphylococcus aureus WCUH29 | Aleatoriu | Clone | Secvențierea | 2.600 | n / A | 168 | n / A | b, c | |
| Staphylococcus aureus RN4220 | Aleatoriu | Clone | Secvențierea | 2.892 | n / A | 658 | 23% | --- | |
| Haemophilus influenzae Rd | Aleatoriu | Populația | Footprint-PCR | 1.657 | 602 | 670 | 40% | --- | |
| Streptococcus pneumoniae Rx-1 | Țintit | Clone | Formarea coloniei | 2.043 | 234 | 113 | n / A | c | |
| Streptococcus pneumoniae D39 | Țintit | Clone | Formarea coloniei | 2.043 | 560 | 133 | n / A | c | |
| Streptococcus pyogenes 5448 | Aleatoriu | Transpozon | Tn-seq | 1.865 | ? | 227 | 12% | --- | |
| Streptococcus pyogenes NZ131 | Aleatoriu | Transpozon | Tn-seq | 1.700 | ? | 241 | 14% | --- | |
| Streptococcus sanguinis SK36 | Țintit | Clone | Formarea coloniei | 2.270 | 2.052 | 218 | 10% | a, j | |
| Mycobacterium tuberculosis H37Rv | Aleatoriu | Populația | Microarray | 3.989 | 2.567 | 614 | 15% | --- | |
| Mycobacterium tuberculosis | Aleatoriu | Transpozon | ? | 3.989 | ? | 401 | 10% | --- | |
| Mycobacterium tuberculosis H37Rv | Aleatoriu | Transpozon | Secvențierea NG | 3.989 | ? | 774 | 19% | --- | |
| Mycobacterium tuberculosis H37Rv | Aleatoriu | Transpozon | Secvențierea NG | 3.989 | 3.364 | 625 | 16% | Salut | |
| Mycobacterium tuberculosis | --- | Computațional | Computațional | 3.989 | ? | 283 | 7% | --- | |
| Bacillus subtilis 168 | Țintit | Clone | Formarea coloniei | 4.105 | 3.830 | 261 | 7% | a, d, g | |
| Escherichia coli K-12 MG1655 | Aleatoriu | Populația | Footprint-PCR | 4.308 | 3.126 | 620 | 14% | --- | |
| Escherichia coli K-12 MG1655 | Țintit | Clone | Formarea coloniei | 4.308 | 2.001 | n / A | n / A | a, e | |
| Escherichia coli K-12 BW25113 | Țintit | Clone | Formarea coloniei | 4.390 | 3.985 | 303 | 7% | A | |
| Pseudomonas aeruginosa PAO1 | Aleatoriu | Clone | Secvențierea | 5.570 | 4.783 | 678 | 12% | A | |
| Porphyromonas gingivalis | Aleatoriu | Transpozon | Secvențierea | 1.990 | 1.527 | 463 | 23% | --- | |
| Pseudomonas aeruginosa PA14 | Aleatoriu | Clone | Secvențierea | 5.688 | 4.469 | 335 | 6% | a, f | |
| Salmonella typhimurium | Aleatoriu | Clone | Secvențierea | 4.425 | n / A | 257 | ~ 11% | b, c | |
| Helicobacter pylori G27 | Aleatoriu | Populația | Microarray | 1.576 | 1.178 | 344 | 22% | --- | |
| Corynebacterium glutamicum | Aleatoriu | Populația | ? | 3.002 | 2.352 | 650 | 22% | --- | |
| Francisella novicida | Aleatoriu | Transpozon | ? | 1.719 | 1.327 | 392 | 23% | --- | |
| Mycoplasma pulmonis UAB CTIP | Aleatoriu | Transpozon | ? | 782 | 472 | 310 | 40% | --- | |
| Vibrio cholerae N16961 | Aleatoriu | Transpozon | ? | 3.890 | ? | 779 | 20% | --- | |
| Salmonella Typhi | Aleatoriu | Transpozon | ? | 4.646 | ? | 353 | 8% | --- | |
| Staphylococcus aureus | Aleatoriu | Transpozon | ? | ~ 2.600 | ? | 351 | 14% | --- | |
| Caulobacter crescentus | Aleatoriu | Transpozon | Tn-Seq | 3.876 | 3.240 | 480 | 12,2% | --- | |
| Neisseria meningitidis | Aleatoriu | Transpozon | ? | 2.158 | ? | 585 | 27% | --- | |
| Desulfovibrio alaskensis | Aleatoriu | Transpozon | Secvențierea | 3.258 | 2.871 | 387 | 12% | --- |
Tabelul 1. Genele esențiale ale bacteriilor . Mutageneză : mutanții vizați sunt deleții genetice; mutanții aleatori sunt inserții de transpozoni . Metode : Clonele indică deleții cu o singură genă, populația indică mutageneză întreagă a populației, de exemplu folosind transpozoni. Genele esențiale din ecranele populației includ gene esențiale pentru fitness (vezi textul). ORF-uri : numărul tuturor cadrelor de citire deschise din genomul respectiv. Note : (a) colecție de mutanți disponibilă; (b) metoda de screening a esențialității directe (de exemplu, prin ARN antisens) care nu oferă informații despre gene neesențiale. (c) Numai set de date parțial disponibil. (d) Include esențialitatea genică prevăzută și compilarea datelor din studii publicate de esențialitate cu o singură genă. (e) Proiect în derulare. (f) Dedus prin compararea celor două seturi de date de esențialitate genică obținute independent în tulpinile P. aeruginosa PA14 și PAO1. (g) Rezultatul inițial al celor 271 de gene esențiale a fost corectat la 261, 31 de gene despre care se credea că sunt esențiale fiind de fapt neesențiale, în timp ce 20 de gene esențiale noi au fost descrise de atunci. (h) Numărarea genelor cu domenii esențiale și a celor care duc la defecte de creștere atunci când sunt perturbate ca fiind esențiale și a celor care conduc la avantaje de creștere atunci când sunt perturbate ca neesențiale. (i) A implicat o bibliotecă mutantă complet saturată de 14 replici, cu 84,3% din site-urile de inserție posibile cu cel puțin o inserție de transpozon. (j) Fiecare genă esențială a fost confirmată independent de cel puțin cinci ori.

Pe baza studiilor experimentale la nivelul genomului și a analizei biologiei sistemelor, o bază de date esențială a genelor a fost dezvoltată de Kong și colab. (2019) pentru prezicerea> 4000 de specii bacteriene.
Eucariote
În Saccharomyces cerevisiae (drojdie în devenire) 15-20% din toate genele sunt esențiale. În Schizosaccharomyces pombe (drojdie de fisiune) au fost construite 4.836 de deleții heterozigote care acoperă 98,4% din cele 4.914 proteine care codifică cadrele de citire deschise. 1.260 dintre aceste ștergeri s-au dovedit a fi esențiale.
Ecranele similare sunt mai dificil de realizat la alte organisme multicelulare, inclusiv la mamifere (ca model pentru oameni), din motive tehnice, iar rezultatele lor sunt mai puțin clare. Cu toate acestea, au fost dezvoltate diverse metode pentru viermele nematod C. elegans , musca fructelor și peștele zebră (vezi tabelul). Un studiu recent realizat pe 900 de gene de șoarece a concluzionat că 42% dintre ele erau esențiale, deși genele selectate nu erau reprezentative.
Experimentele de eliminare a genelor nu sunt posibile sau cel puțin nu sunt etice la om. Cu toate acestea, mutațiile naturale au condus la identificarea mutațiilor care duc la moartea embrionară timpurie sau mai târziu. Rețineți că multe gene la om nu sunt absolut esențiale pentru supraviețuire, dar pot provoca boli severe atunci când sunt mutate. Astfel de mutații sunt catalogate în baza de date Online Mendelian Inheritance in Man (OMIM). Intr - o analiza computațională de variatie genetica si mutatii in 2472 umane ortologi de gene esentiale cunoscute in mouse - ul, Georgi și colab. au găsit o selecție puternică, purificatoare și niveluri relativ reduse de variație a secvenței, indicând că și aceste gene umane sunt esențiale.
Deși poate fi dificil să se demonstreze că o genă este esențială la om, se poate demonstra că o genă nu este esențială sau chiar nu provoacă boli. De exemplu, secvențierea genomului a 2.636 de cetățeni islandezi și genotiparea a 101.584 subiecți suplimentari au găsit 8.041 de indivizi care aveau o genă complet eliminată (adică acești oameni erau homozigoți pentru o genă nefuncțională). Dintre cei 8.041 de indivizi cu knock-out complet, 6.885 s-au estimat a fi homozigoti , 1.249 s-au estimat a fi heterozigoți compuși (adică aveau ambele alele ale unei gene eliminate, dar cele două alele aveau mutații diferite). La acești indivizi, un total de 1.171 din 19.135 gene umane ( RefSeq ) (6,1%) au fost complet eliminate. S-a ajuns la concluzia că aceste 1.171 de gene sunt neesențiale la om - cel puțin nu au fost raportate boli asociate. În mod similar, secvențele exome ale a 3222 de adulți britanici cu moștenire pakistaneză cu relații parentale ridicate au dezvăluit 1111 genotipuri homozigote cu variante rare cu pierderea prezisă a funcției genice (LOF = knockouts) la 781 de gene. Acest studiu a găsit o medie de 140 de genotipuri LOF prevăzute (pe subiect), incluzând 16 heterozigoți rari ( frecvență alelă minoră <1%), 0,34 homozigoți rari, 83,2 heterozigoți comuni și 40,6 homozigoți comuni. Aproape toate genotipurile LOF homozigote rare au fost găsite în segmentele autozigote (94,9%). Chiar dacă majoritatea acestor indivizi nu au avut nicio problemă evidentă de sănătate care rezultă din genele lor defecte, este posibil ca problemele minore de sănătate să poată fi găsite la o examinare mai detaliată.
Un rezumat al ecranelor de esențialitate este prezentat în tabelul de mai jos (în principal bazat pe baza de date a genelor esențiale.
| Organism | Metodă | Gene esențiale | Ref. |
| Arabidopsis thaliana | Inserarea ADN-ului T | 777 | |
| Caenorhabditis elegans (vierme) | Interferența ARN | 294 | |
| Danio rerio ( pește zebră) | Mutageneză de inserție | 288 | |
| Drosophila melanogaster (musca fructelor) | Mutageneză de inserție a elementului P | 339 | |
| Homo sapiens (uman) | Căutare literatură | 118 | |
| Homo sapiens (uman) | Ecran bazat pe CRISPR / Cas9 | 1.878 | |
| Homo sapiens (uman) | Ecranul capcanei genetice haploide | ~ 2.000 | |
| Homo sapiens (uman) | ortologi de șoarece | 2.472 | |
| Mus musculus (șoarece) | Căutare literatură | 2114 | |
| Saccharomyces cerevisiae (drojdie) | Deleții cu o singură genă | 878 | |
| Saccharomyces cerevisiae (drojdie) | Deleții cu o singură genă | 1.105 | |
| Schizosaccharomyces pombe (drojdie) | Deleții cu o singură genă | 1.260 |
Viruși
Virușilor le lipsesc multe gene necesare metabolismului, forțându-i să deturneze metabolismul gazdei. Au fost efectuate ecrane pentru gene esențiale în câțiva viruși. De exemplu, sa constatat că citomegalovirusul uman (CMV) are 41 de ORF-uri esențiale, 88 neesențiale și 27 (150 ORF-uri totale). Cele mai multe gene esențiale și crescătoare sunt situate în regiunea centrală, iar genele neesențiale se îngrămădesc în general lângă capetele genomului viral.
Tscharke și Dobson (2015) au compilat un studiu cuprinzător al genelor esențiale în virusul Vaccinia și au atribuit roluri fiecăruia dintre cele 223 ORF ale tulpinii Western Reserve (WR) și 207 ORF ale tulpinii Copenhaga, evaluând rolul lor în replicarea în cultura celulară. Conform definiției lor, o genă este considerată esențială (adică are un rol în cultura celulară) dacă ștergerea acesteia are ca rezultat o scădere a titrului virusului de peste 10 ori, fie într-o curbă de creștere cu un singur pas, fie cu mai multe etape. Toate genele implicate în producția de virion înfășurat, formarea cozii de actină și eliberarea extracelulară de virion au fost, de asemenea, considerate esențiale. Genele care influențează dimensiunea plăcii, dar nu replicarea, au fost definite ca neesențiale. Prin această definiție, 93 de gene sunt necesare pentru replicarea virusului Vaccinia în cultura celulară, în timp ce 108 și 94 ORF, de la WR și respectiv de la Copenhaga, sunt neesențiale. Virușii Vaccinia cu deleții la fiecare capăt al genomului s-au comportat așa cum era de așteptat, prezentând doar defecte ușoare sau ale distanței gazdei. În schimb, combinarea ștergerilor la ambele capete ale genomului pentru tulpina VACV WR a provocat un defect de creștere devastator pe toate liniile celulare testate. Acest lucru demonstrează că eliminările unei singure gene nu sunt suficiente pentru a evalua esențialitatea genelor și că mai multe gene sunt esențiale în virusul Vaccinia decât se credea inițial.
Unul dintre bacteriofagii selectați pentru gene esențiale include micobacteriofagul Giles. Cel puțin 35 din cele 78 de gene Giles prezise (45%) sunt neesențiale pentru creșterea litică. 20 de gene s-au dovedit a fi esențiale. O problemă majoră a genelor fagice este că majoritatea genelor lor rămân necunoscute funcțional, prin urmare rolul lor este dificil de evaluat. Un ecran al fagului Salmonella enterica SPN3US a dezvăluit 13 gene esențiale, deși rămâne puțin obscur câte gene au fost testate cu adevărat.
Analiza cantitativă a esențialității genelor
În teorie, genele esențiale sunt calitative. Cu toate acestea, în funcție de mediul înconjurător, anumite mutante genetice esențiale pot prezenta funcții parțiale, care pot fi determinate cantitativ în unele studii. De exemplu, o anumită ștergere a genei poate reduce rata de creștere (sau rata fertilității sau alte caractere) la 90% din tipul sălbatic. Dacă există izozime sau căi alternative pentru genele esențiale, acestea pot fi șterse complet.
Letalitate sintetică
Două gene sunt sintetice letale dacă niciuna dintre ele nu este esențială, dar atunci când ambele sunt mutante, dublul mutant este letal. Unele studii au estimat că numărul de gene letale sintetice poate fi de ordinul a 45% din toate genele.
Gene esențiale condiționate

Multe gene sunt esențiale numai în anumite circumstanțe. De exemplu, dacă aminoacidul lizină este furnizat unei celule, orice genă care este necesară pentru a produce lizina este neesențială. Cu toate acestea, atunci când nu este furnizată lizină, genele care codifică enzime pentru biosinteza lizinei devin esențiale, deoarece nu este posibilă sinteza proteinelor fără lizină.
Streptococcus pneumoniae pare să necesite 147 de gene pentru creșterea și supraviețuirea salivei , mai mult decât 113-133 care au fost găsite în studiile anterioare.
Ștergerea unei gene poate duce la moarte sau la blocarea diviziunii celulare . În timp ce ultimul caz poate implica „supraviețuirea” pentru o perioadă de timp, fără divizarea celulară, celula poate muri în cele din urmă. În mod similar, în loc de blocarea diviziunii celulare, o celulă poate avea o creștere sau un metabolism redus , de la aproape nedetectabil la aproape normal. Astfel, există un gradient de la „esențial” la complet neesențial, din nou în funcție de condiție. Unii autori au distins astfel între gene „ esențiale pentru supraviețuire ” și „ esențiale pentru fitness ”.
Rolul fundalului genetic . Similar condițiilor de mediu, fundalul genetic poate determina esențialitatea unei gene: o genă poate fi esențială la un individ, dar nu la altul, având în vedere originea sa genetică. Duplicările genetice sunt o explicație posibilă (vezi mai jos).
Dependența metabolică . Genele implicate în anumite căi biosintetice, cum ar fi sinteza aminoacizilor , pot deveni neesențiale dacă unul sau mai mulți aminoacizi sunt furnizați de mediul de cultură sau de un alt organism. Acesta este principalul motiv pentru care mulți paraziți (de exemplu, Cryptosporidium hominis ) sau bacterii endosimbiontice au pierdut multe gene (de exemplu, Chlamydia ). Astfel de gene pot fi esențiale, dar prezente doar în organismul gazdă. De exemplu, Chlamydia trachomatis nu poate sintetiza nucleotide purine și pirimidine de novo , astfel încât aceste bacterii sunt dependente de genele nucleotidice biosintetice ale gazdei.
Un alt tip de dependență metabolică, fără legătură cu interacțiunile între specii, poate fi găsit atunci când bacteriile sunt cultivate în condiții specifice de nutrienți . De exemplu, mai mult de 100 de gene devin esențiale atunci când Escherichia coli este cultivată pe un mediu limitat de nutrienți. Mai exact, izocitrat dehidrogenază ( icd ) și citrat sintază (gltA) sunt două enzime care fac parte din ciclul acidului tricarboxilic (TCA) . Ambele gene sunt esențiale în mediile minime M9 (care oferă doar substanțele nutritive de bază). Cu toate acestea, atunci când mediul suplimentează cu 2-oxoglutarat sau glutamat , aceste gene nu mai sunt esențiale.
Duplicări genetice și căi metabolice alternative
Multe gene sunt duplicate într-un genom și multe organisme au căi metabolice diferite (cale metabolică alternativă) pentru a sintetiza aceleași produse. Astfel de duplicări ( paralogi ) și căi metabolice alternative fac adesea gene esențiale neesențiale, deoarece duplicatul poate înlocui copia originală. De exemplu, gena care codifică enzima aspartokinază este esențială în E. coli . Prin contrast, genomul Bacillus subtilis conține trei copii ale acestei gene, dintre care niciuna nu este esențială. Cu toate acestea, o triplă ștergere a tuturor celor trei gene este letală. În astfel de cazuri, esențialitatea unei gene sau a unui grup de paralogi poate fi adesea prezisă pe baza esențialității unei gene esențiale unice la o specie diferită. În drojdie, puține dintre genele esențiale sunt duplicate în genom: 8,5% din genele neesențiale, dar doar 1% din genele esențiale au un omolog în genomul drojdiei.
La viermele C. elegans , genele neesențiale sunt extrem de supra-reprezentate printre duplicate, posibil, deoarece duplicarea genelor esențiale determină supraexprimarea acestor gene. Woods și colab. a constatat că genele neesențiale sunt mai des duplicate cu succes (fixate) și pierdute în comparație cu genele esențiale. Prin contrast, genele esențiale sunt mai rar duplicate, dar după o duplicare reușită sunt menținute pe perioade mai lungi.
Conservare
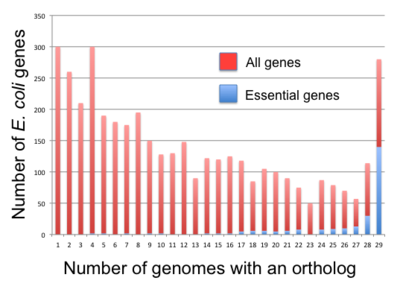
La bacterii , genele esențiale par a fi mai conservate decât genele neesențiale, dar corelația nu este foarte puternică. De exemplu, doar 34% din genele esențiale B. subtilis au ortologi de încredere în toate Firmicutes și 61% din genele esențiale de E. coli au ortologi de încredere în toate gamma-proteobacteriile . Fang și colab. (2005) au definit genele persistente ca fiind genele prezente în mai mult de 85% din genomurile cladei. Au descoperit 475 și 611 din astfel de gene pentru B. subtilis și , respectiv, pentru E. coli . Mai mult, au clasificat genele în cinci clase în funcție de persistență și esențialitate: gene persistente, gene esențiale, gene persistente neesențiale (PNE) (276 în B. subtilis , 409 în E. coli ), gene esențiale non-persistente (ENP) (73 în B . subtilis , 33 în E. coli ) și gene non-persistente neesențiale (NPNE) (3.558 în B. subtilis , 3.525 în E. coli ). Fang și colab. au găsit 257 de gene persistente, care există atât în B. subtilis (pentru Firmicutes), cât și în E. coli (pentru Gamma-proteobacteria). Dintre acestea, 144 (respectiv 139) au fost identificate anterior ca fiind esențiale în B. subtilis (respectiv E. coli ) și 25 (respectiv 18) din cele 257 de gene nu sunt prezente în 475 B. subtilis (respectiv 611 E. coli) persistente gene. Toți ceilalți membri ai grupului sunt gene PNE.
În eucariote , 83% dintre ortologii individuali între Schizosaccharomyces pombe și Saccharomyces cerevisiae au păstrat esențialitatea, adică sunt neesențiale la ambele specii sau esențiale la ambele specii. Restul de 17% din gene sunt neesențiale la o specie și esențiale la cealaltă. Acest lucru este destul de remarcabil, având în vedere că S. pombe este separată de S. cerevisiae cu aproximativ 400 de milioane de ani de evoluție.
În general, genele foarte conservate și, prin urmare, mai vechi (adică genele cu origine filogenetică anterioară) sunt mai susceptibile de a fi esențiale decât genele mai tinere - chiar dacă au fost duplicate.
Studiu
Studiul experimental al genelor esențiale este limitat de faptul că, prin definiție, inactivarea unei gene esențiale este letală pentru organism. Prin urmare, ele nu pot fi pur și simplu șterse sau mutate pentru a analiza fenotipurile rezultate (o tehnică comună în genetică ).
Există, totuși, unele circumstanțe în care genele esențiale pot fi manipulate. La organismele diploide , poate fi necesară doar o singură copie funcțională a unor gene esențiale ( haplosuficiență ), heterozigotul prezentând un fenotip instructiv. Unele gene esențiale pot tolera mutații care sunt dăunătoare, dar nu total letale, deoarece nu abolesc complet funcția genei.
Analiza computațională poate dezvălui multe proprietăți ale proteinelor fără a le analiza experimental, de exemplu, analizând proteinele omoloage , funcția, structura etc. (vezi și mai jos, Predicția genelor esențiale ). Produsele genelor esențiale pot fi, de asemenea, studiate atunci când sunt exprimate în alte organisme sau când sunt purificate și studiate in vitro .
Genele esențiale condiționate sunt mai ușor de studiat. Au fost identificate variante sensibile la temperatură ale genelor esențiale care codifică produsele care își pierd funcția la temperaturi ridicate și, prin urmare, prezintă doar un fenotip la temperatură crescută.
Reproductibilitate
Dacă ecranele pentru gene esențiale sunt repetate în laboratoare independente, acestea duc adesea la liste diferite de gene. De exemplu, ecranele din E. coli au dat de la 300 la ~ 600 gene esențiale (vezi Tabelul 1 ). Astfel de diferențe sunt și mai accentuate atunci când se utilizează diferite tulpini bacteriene (vezi Figura 2 ). O explicație comună este că condițiile experimentale sunt diferite sau că natura mutației poate fi diferită (de exemplu, o ștergere completă a genei vs. un mutant transposon). Ecranele cu transpozoni în special sunt greu de reprodus, având în vedere că un transposon se poate insera în mai multe poziții în cadrul unei gene. Inserțiile către capătul 3 'al unei gene esențiale pot să nu aibă un fenotip letal (sau deloc fenotip) și, prin urmare, nu pot fi recunoscute ca atare. Acest lucru poate duce la adnotări eronate (aici: negative negative).
Compararea ecranelor CRISPR / cas9 și RNAi . Ecranele pentru identificarea genelor esențiale din linia celulară K562 de leucemie mielogenă cronică umană cu aceste două metode au arătat doar o suprapunere limitată. La o rată fals pozitivă de 10%, au existat ~ 4.500 de gene identificate în ecranul Cas9 comparativ cu ~ 3.100 în ecranul shRNA , cu doar ~ 1.200 gene identificate în ambele.
Diferite gene esențiale în diferite organisme
Diferite organisme pot avea gene esențiale diferite. De exemplu, Bacillus subtilis are 271 de gene esențiale. Aproximativ jumătate (150) din genele ortoloage din E. coli sunt, de asemenea, esențiale. Alte 67 de gene care sunt esențiale în E. coli nu sunt esențiale în B. subtilis , în timp ce 86 de gene E. esențiale nu au ortolog B. subtilis . La Mycoplasma genitalium sunt esențiale cel puțin 18 gene care nu sunt esențiale la M. bovis. Multe dintre aceste gene esențiale diferite sunt cauzate de paralogi sau căi metabolice alternative.
Astfel de gene esențiale diferite ale bacteriilor pot fi utilizate pentru a dezvolta terapii antibacteriene vizate împotriva anumitor agenți patogeni specifici pentru a reduce rezistența la antibiotice în era microbiomului. Stone și colab. (2015) au folosit diferența de gene esențiale în bacterii pentru a dezvolta medicamente selective împotriva agentului patogen oral Porphyromonas gingivalis , mai degrabă decât bacteriile benefice Streptococcus sanguis .
Predicție
Genele esențiale pot fi prezise prin calcul. Cu toate acestea, majoritatea metodelor folosesc într-o oarecare măsură date experimentale („seturi de antrenament”). Chen și colab. a stabilit patru criterii pentru a selecta seturi de antrenament pentru astfel de predicții: (1) genele esențiale din setul de antrenament selectat ar trebui să fie fiabile; (2) condițiile de creștere în care sunt definite genele esențiale ar trebui să fie consecvente în seturile de instruire și predicție; (3) speciile utilizate ca set de antrenament ar trebui să fie strâns legate de organismul țintă; și (4) organismele utilizate ca seturi de antrenament și predicție ar trebui să prezinte fenotipuri sau stiluri de viață similare. Ei au descoperit, de asemenea, că dimensiunea setului de antrenament ar trebui să fie de cel puțin 10% din totalul genelor pentru a produce predicții precise. Unele abordări pentru prezicerea genelor esențiale sunt:
Genomică comparativă . La scurt timp după ce primele genome (ale Haemophilus influenzae și Mycoplasma genitalium ) au devenit disponibile, Mushegian și colab. a încercat să prezică numărul de gene esențiale bazate pe gene comune la aceste două specii. S-a presupus că numai genele esențiale ar trebui conservate pe distanța evolutivă mare care a separat cele două bacterii. Acest studiu a identificat aproximativ 250 de gene esențiale candidate. Pe măsură ce mai multe genome au devenit disponibile, numărul de gene esențiale prevăzute a continuat să se micșoreze, deoarece mai multe genome au împărțit din ce în ce mai puține gene. În consecință, s-a ajuns la concluzia că nucleul universal conservat este format din mai puțin de 40 de gene. Cu toate acestea, acest set de gene conservate nu este identic cu setul de gene esențiale, deoarece diferite specii se bazează pe gene esențiale diferite.
O abordare similară a fost folosită pentru a deduce gene esentiale din pan-genomului de Brucella specii. 42 de genomi Brucella complet și un total de 132.143 de gene care codifică proteinele au fost folosite pentru a prezice 1252 de gene esențiale potențiale, derivate din genomul de bază prin comparație cu o bază de date procariote de gene esențiale.
Analiza rețelei . După ce au fost publicate primele rețele de interacțiune cu drojdie , s-a constatat că proteinele extrem de conectate (de exemplu, prin interacțiuni proteină-proteină ) sunt mai susceptibile de a fi esențiale. Cu toate acestea, proteinele extrem de conectate pot fi artefacte experimentale, iar conectivitatea ridicată poate reprezenta mai degrabă pleiotropie în loc de esențialitate. Cu toate acestea, metodele de rețea au fost îmbunătățite prin adăugarea altor criterii și, prin urmare, au o anumită valoare în prezicerea genelor esențiale.
Învățarea automată . Hua și colab. a folosit Machine Learning pentru a prezice gene esențiale la 25 de specii bacteriene .
Indicele Hurst . Liu și colab. (2015) au folosit exponentul Hurst , un parametru caracteristic pentru a descrie corelația pe termen lung în ADN pentru a prezice genele esențiale. La 31 din 33 de genomi bacterieni, nivelurile de semnificație ale exponenților Hurst ai genelor esențiale au fost semnificativ mai mari decât pentru setul de gene complet corespunzător, în timp ce nivelurile de semnificație ale exponenților Hurst ai genelor neesențiale au rămas neschimbate sau au crescut doar ușor.
Genomi minimi . De asemenea, s-a crezut că genele esențiale ar putea fi deduse din genomuri minime care presupun că conțin doar gene esențiale. Problema aici este că cele mai mici genomi aparțin speciilor parazite (sau simbiontice) care pot supraviețui cu un set de gene redus, deoarece obțin mulți nutrienți de la gazdele lor. De exemplu, unul dintre cele mai mici genomi este cel al Hodgkinia cicadicola , un simbiont de cicade, care conține doar 144 Kb de ADN care codifică doar 188 de gene. Ca și alți simbionți, Hodgkinia primește multe dintre substanțele nutritive de la gazda sa, astfel încât genele sale nu trebuie să fie esențiale.
Modelarea metabolică . Genele esențiale pot fi, de asemenea, prezise în genomuri complet secvențiate prin reconstrucție metabolică , adică prin reconstituirea metabolismului complet din conținutul genei și apoi identificarea acelor gene și căi care s-au dovedit a fi esențiale la alte specii. Cu toate acestea, această metodă poate fi compromisă de proteinele cu funcție necunoscută. În plus, multe organisme au căi de rezervă sau alternative care trebuie luate în considerare (a se vedea figura 1). Modelarea metabolică a fost, de asemenea, utilizată de Basler (2015) pentru a dezvolta o metodă de predicție a genelor metabolice esențiale. Analiza echilibrului fluxului , o metodă de modelare metabolică, a fost recent utilizată pentru a prezice genele esențiale în metabolismul carcinomului cu celule renale cu celule clare.
Gene de funcție necunoscută . În mod surprinzător, un număr semnificativ de gene esențiale nu are nicio funcție cunoscută. De exemplu, dintre cei 385 de candidați esențiali din M. genitalium , nicio funcție nu putea fi atribuită la 95 de gene, chiar dacă acest număr fusese redus la 75 până în 2011. Majoritatea genelor funcționale esențiale necunoscute au funcții biologice potențiale legate de una dintre cele trei fundamentale. funcții.
ZUPLS . Song și colab. a prezentat o metodă nouă pentru a prezice gene esențiale care utilizează numai curba Z și alte caracteristici bazate pe secvență. Astfel de caracteristici pot fi calculate ușor din secvențele ADN / aminoacizi. Cu toate acestea, fiabilitatea acestei metode rămâne puțin obscură.
Servere esențiale de predicție a genelor . Guo și colab. (2015) au dezvoltat trei servicii online pentru a prezice genele esențiale din genomul bacterian. Aceste instrumente disponibile gratuit sunt aplicabile pentru secvențe cu o singură genă fără funcții adnotate, gene cu nume definite și genomuri complete ale tulpinilor bacteriene. Kong și colab. (2019) au dezvoltat baza de date ePath , care poate fi utilizată pentru a căuta> 4000 de specii bacteriene pentru a prezice gene esențiale.
Domenii esențiale de proteine
Deși majoritatea genelor esențiale codifică proteinele, multe proteine esențiale constau dintr-un singur domeniu. Acest fapt a fost folosit pentru a identifica domeniile proteice esențiale. Goodacre și colab. au identificat sute de domenii esențiale cu funcție necunoscută (eDUF). Lu și colab. au prezentat o abordare similară și au identificat 3.450 de domenii care sunt esențiale la cel puțin o specie microbiană.
Vezi si
Referințe
Lecturi suplimentare
- Gao F, Luo H, Zhang CT, Zhang R (2015). "Analiza esențialității genelor bazată pe DEG 10, o bază de date actualizată a genelor esențiale". Esențialitatea genei . Metode în biologie moleculară. 1279 . pp. 219–33. doi : 10.1007 / 978-1-4939-2398-4_14 . ISBN 978-1-4939-2397-7. PMID 25636622 .
- Long JL, ed. (2015). Esențialitatea genelor - Metode și protocoale Springer . Metode în biologie moleculară. 1279 . Humana Press. p. 248. doi : 10.1007 / 978-1-4939-2398-4 . ISBN 978-1-4939-2397-7. S2CID 27547825 .
linkuri externe
- Baza de date a genelor esențiale
- OGEE: Baza de date online esențială
- Bază de date EGGS (Essential Genes on Genome Scale)
- Baza de date ePath (Gene esențiale în cale)
- Gene esențiale în E. coli (EcoliWiki )
- Gene esențiale în E. coli (Ecogene)
- Genele esențiale ale lui Benjamin Lewin (manual), Pearson / Prentice-Hall .
