Splicing alternativ - Alternative splicing

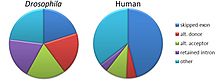
Splicarea alternativă sau splicarea alternativă a ARN-ului sau splicarea diferențială este un proces alternativ de splicare în timpul exprimării genei care permite unei singure gene să codifice mai multe proteine . În acest proces, exoni particulari ai unei gene pot fi incluși în sau excluși din ARN-ul mesager final (ARNm) procesat produs din acea genă. Acest lucru înseamnă că exonii sunt uniți în diferite combinații, ducând la diferite catene de ARNm (alternative). În consecință, proteinele traduse din ARNm cu splicing alternativ vor conține diferențe în secvența lor de aminoacizi și, adesea, în funcțiile lor biologice (vezi Figura). În special, îmbinarea alternativă permite genomului uman să dirijeze sinteza multor mai multe proteine decât s-ar aștepta de la cele 20.000 de gene care codifică proteinele.
Împletirea alternativă apare ca un fenomen normal în eucariote , unde crește foarte mult biodiversitatea proteinelor care pot fi codificate de genom; la om, ~ 95% din genele multi-exonice sunt alternativ îmbinate. Există numeroase moduri de îmbinare alternativă observate, dintre care cel mai frecvent este săritul exonului . În acest mod, un anumit exon poate fi inclus în ARNm în anumite condiții sau în anumite țesuturi și omis din ARNm în altele.
Producția de mARN alternativ îmbinată este reglementată de un sistem de proteine cu acțiune trans care se leagă de siturile cu acțiune cis pe transcriptul primar în sine. Astfel de proteine includ activatori de îmbinare care promovează utilizarea unui anumit sit de îmbinare și represori de îmbinare care reduc utilizarea unui anumit sit. Mecanismele de îmbinare alternativă sunt extrem de variabile și se găsesc în mod constant noi exemple, în special prin utilizarea tehnicilor de mare viteză. Cercetătorii speră să elucideze pe deplin sistemele de reglementare implicate în splicing, astfel încât produsele alternative de splicing dintr-o anumită genă în anumite condiții („variante splicing”) să poată fi prezise printr-un „cod splicing”.
Variațiile anormale ale îmbinării sunt implicate și în boală ; o proporție mare de tulburări genetice umane rezultă din variantele de îmbinare. Se consideră, de asemenea, că variante anormale de îmbinare contribuie la dezvoltarea cancerului, iar genele factorului de îmbinare sunt frecvent mutate în diferite tipuri de cancer.
Descoperire
Splicingul alternativ a fost observat pentru prima dată în 1977. Adenovirusul produce cinci transcrieri primare la începutul ciclului său infecțios, înainte de replicarea ADN-ului viral, și unul suplimentar mai târziu, după începerea replicării ADN-ului. Transcrierile primare primare continuă să fie produse după începerea replicării ADN-ului. Transcriptul primar suplimentar produs târziu în infecție este mare și provine de la 5/6 din genomul adenovirusului de 32kb. Acesta este mult mai mare decât oricare dintre mARN-urile adenovirusului individuale prezente în celulele infectate. Cercetătorii au descoperit că transcriptul primar de ARN produs de adenovirus de tip 2 în faza târzie a fost îmbinat în multe moduri diferite, rezultând ARNm care codifică diferite proteine virale. În plus, transcrierea primară conținea mai multe situri de poliadenilare , oferind capete 3 'diferite pentru mARN-urile procesate.
În 1981, a fost caracterizat primul exemplu de îmbinare alternativă într-un transcript dintr-o genă normală, endogenă . S- a constatat că gena care codifică calcitonina hormonului tiroidian este îmbinată alternativ în celulele mamiferelor. Transcriptul primar din această genă conține 6 exoni; calcitonina ARNm conține exonii 1-4, și se termină după un poliadenilare sit în exonul 4. Un alt ARNm este produs din această pre-ARNm sărind peste exonul 4 și include exonii 1-3, 5 și 6. codifică o proteină cunoscută ca CGRP ( peptida legată de gena calcitoninei ). Exemple de splicing alternativ în transcrierile genei imunoglobinei la mamifere au fost, de asemenea, observate la începutul anilor 1980.
De atunci, splicarea alternativă sa dovedit a fi omniprezentă în eucariote. "Deținătorul recordului" pentru îmbinarea alternativă este o genă D. melanogaster numită Dscam , care ar putea avea 38.016 variante de îmbinare.
În 2021, s-a descoperit că genomul adenovirusului de tip 2, adenovirusul în care s-a identificat mai întâi splicingul alternativ, a putut produce o varietate mult mai mare de ARNm decât se credea anterior. Prin utilizarea tehnologiei de secvențiere a generației următoare, cercetătorii au reușit să actualizeze transcriptomul de tip 2 al adenovirusului uman și să prezinte un ARNm 904 unic, uluitor, produs de virus printr-un model complex de îmbinare alternativă.
Moduri
Cinci moduri de bază de îmbinare alternativă sunt în general recunoscute.
- Saltarea exonului sau exonul casetei : în acest caz, un exon poate fi splicat din transcrierea primară sau reținut. Acesta este cel mai comun mod la pre-ARNm la mamifere .
- Exoni care se exclud reciproc : unul dintre cei doi exoni este reținut în ARNm după îmbinare, dar nu în ambii.
- Site donator alternativ : Se utilizează o joncțiune alternativă de 5 '(sit donator), schimbând limita de 3' a exonului din amonte .
- Site acceptor alternativ : Se utilizează o joncțiune de 3 'splice alternativă (site acceptor), schimbând limita 5' a exonului din aval.
- Retenția intronului: o secvență poate fi îmbinată ca un intron sau pur și simplu reținută. Acest lucru se distinge de saltarea exonului, deoarece secvența reținută nu este flancată de introni . Dacă intronul reținut se află în regiunea de codificare, intronul trebuie să codifice aminoacizii în cadru cu exonii vecini, sau un codon de oprire sau o deplasare în cadrul de citire va face ca proteina să fie nefuncțională. Acesta este cel mai rar mod la mamifere.
În plus față de aceste moduri primare de îmbinare alternativă, există alte două mecanisme principale prin care ARNm diferiți pot fi generați din aceeași genă; promotori multipli și site-uri multiple de poliadenilare . Utilizarea mai multor promotori este descrisă în mod corespunzător ca un mecanism de reglare a transcripției , mai degrabă decât o îmbinare alternativă; pornind transcrierea în diferite puncte, se pot genera transcrieri cu 5'-exoni diferiți. La celălalt capăt, mai multe situri de poliadenilare oferă puncte finale 3 'diferite pentru transcriere. Ambele mecanisme se găsesc în combinație cu îmbinarea alternativă și oferă o varietate suplimentară în ARNm derivate dintr-o genă.

Aceste moduri descriu mecanisme de îmbinare de bază, dar pot fi inadecvate pentru a descrie evenimente complexe de îmbinare. De exemplu, figura din dreapta prezintă 3 forme de splice din gena hialuronidazei 3 de șoarece. Compararea structurii exonice prezentate în prima linie (verde) cu cea din a doua linie (galbenă) arată retenția intronului, în timp ce comparația dintre a doua și a treia formă de îmbinare (galben vs. albastru) prezintă sărituri de exon. Recent a fost propusă o nomenclatură de model care să desemneze în mod unic toate modelele posibile de îmbinare.
Mecanisme
Mecanism general de îmbinare
Când pre-ARNm a fost transcris din ADN , acesta include mai mulți introni și exoni . (La nematode , media este de 4-5 exoni și introni; în mușchiul fructului Drosophila pot exista mai mult de 100 de introni și exoni într-un pre-ARNm transcris.) Exonii care trebuie reținuți în ARNm sunt determinați în timpul procesului de îmbinare . Reglarea și selecția site-urilor de îmbinare se realizează prin activarea trans-acționare splicing și splicing proteine represoare, precum și elemente cu acțiune cis în pre-ARNm în sine, cum ar fi amplificatori explicare splicing și amortizoare exonere amortizoare.
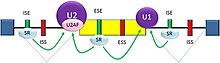
Intronul nuclear eucariot tipic are secvențe de consens care definesc regiuni importante. Fiecare intron are secvența GU la capătul său 5 '. Aproape de capătul 3 'există un site de ramură. Nucleotida la punctul de ramificare este întotdeauna un A; consensul în jurul acestei secvențe variază oarecum. La om secvența consensului site-ului ramurii este yUnAy. Locul ramificării este urmat de o serie de pirimidine - tractul polipirimidinic - apoi de AG la capătul 3 '.
Splicarea ARNm este realizată de un complex de ARN și proteine cunoscut sub numele de spliceozom , conținând snRNP-uri desemnate U1, U2 , U4, U5 și U6 (U3 nu este implicat în splicarea ARNm). U1 se leagă de 5 'GU și U2, cu asistența factorilor de proteină U2AF , se leagă de punctul de ramificare A din cadrul situsului de ramificare. Complexul din această etapă este cunoscut sub numele de complex spliceozom A. Formarea complexului A este de obicei pasul cheie în determinarea capetelor intronului care urmează să fie îmbinat și în definirea capetelor exonului care trebuie păstrat. (Nomenclatura U derivă din conținutul lor ridicat de uridină).
Complexul U4, U5, U6 se leagă și U6 înlocuiește poziția U1. U1 și U4 pleacă. Complexul rămas efectuează apoi două reacții de transesterificare . În prima transesterificare, capătul 5 'al intronului este scindat de exonul din amonte și legat la locul ramificării A printr-o legătură 2', 5'- fosfodiester . În a doua transesterificare, capătul 3 'al intronului este scindat de exonul din aval, iar cei doi exoni sunt uniți printr-o legătură fosfodiester. Intronul este apoi eliberat sub formă de lariat și degradat.
Elemente de reglementare și proteine
Splicing-ul este reglat de proteinele cu acțiune trans (represori și activatori) și de siturile de reglare cu acțiune cis corespunzătoare (amortizoare și amplificatoare) pe pre-ARNm. Cu toate acestea, ca parte a complexității îmbinării alternative, se observă că efectele unui factor de îmbinare sunt frecvent dependente de poziție. Adică, un factor de îmbinare care servește ca activator de îmbinare atunci când este legat de un element de amplificare intronic poate servi ca un represor atunci când este legat de elementul său de îmbinare în contextul unui exon și invers. Structura secundară a transcrierii pre-ARNm joacă, de asemenea, un rol în reglarea splicingului, cum ar fi prin reunirea elementelor de splicing sau prin mascarea unei secvențe care altfel ar servi ca element de legare pentru un factor de splicing. Împreună, aceste elemente formează un „cod de splicing” care guvernează modul în care splicingul va avea loc în diferite condiții celulare.
Există două tipuri majore de elemente de secvență de ARN cu acțiune cis prezente în pre-ARNm și au proteine corespunzătoare care leagă ARN cu acțiune trans . Splicing amortizoare de zgomot sunt site - uri la care proteinele despicare represor se leaga, reducând probabilitatea ca un sit din apropiere va fi folosit ca o joncțiune matisare. Acestea pot fi localizate în intron în sine (amortizoare de zgomot intronic, ISS) sau într-un exon învecinat ( amortizoare de zgomot exonic , ESS). Ele variază în ordine, precum și în tipurile de proteine care se leagă de ele. Majoritatea represorilor de îmbinare sunt ribonucleoproteine nucleare eterogene (hnRNPs), cum ar fi hnRNPA1 și proteina de legare a tractului polipirimidinic (PTB). Amplificatorii de îmbinare sunt site-uri la care se leagă proteinele activatoare de îmbinare, crescând probabilitatea ca un sit din apropiere să fie folosit ca joncțiune de îmbinare. Acestea pot apărea, de asemenea, în intron (amplificatori de îmbinare intronică, ISE) sau exon ( amplificatori de îmbinare exonică , ESE). Majoritatea proteinelor activatoare care se leagă de ISE și ESE sunt membri ai familiei de proteine SR . Astfel de proteine conțin motive de recunoaștere a ARN și domenii bogate în arginină și serină (RS).
În general, factorii determinanți ai splicingului funcționează într-o manieră interdependentă care depinde de context, astfel încât regulile care reglementează modul în care splicing-ul este reglementat dintr-un cod de splicing. Prezența unui anumit element de secvență de ARN cu acțiune cis poate crește probabilitatea ca un site din apropiere să fie îmbinat în unele cazuri, dar să scadă probabilitatea în alte cazuri, în funcție de context. Contextul în care acționează elementele de reglementare include contextul care acționează cis, care este stabilit prin prezența altor caracteristici ale secvenței ARN și contextul care acționează trans, care este stabilit de condițiile celulare. De exemplu, unele elemente de secvență ARN cu acțiune cis influențează îmbinarea numai dacă mai multe elemente sunt prezente în aceeași regiune, astfel încât să se stabilească contextul. Ca un alt exemplu, un element cu acțiune cis poate avea efecte opuse asupra îmbinării, în funcție de proteinele exprimate în celulă (de exemplu, PTB neuronal versus non-neuronal). Semnificația adaptativă a amortizoarelor de izolare și a amplificatorilor este atestată de studii care arată că există o selecție puternică în genele umane împotriva mutațiilor care produc noi amortizoare de zgomot sau care perturbă amplificatoarele existente.
Metilarea ADN-ului și îmbinarea alternativă la insectele sociale
Metilarea ADN-ului CpG a arătat un rol de a regla îmbinarea alternativă la insectele sociale. La albinele de miere ( Apis mellifera ), metilarea ADN-ului CpG pare să regleze saltarea exonului pe baza primelor studii genomice după ce genomul albinei a fost disponibil. Metilarea ADN-ului CpG a reglementat îmbinarea alternativă mai extinsă, nu numai că afectează sărirea exonului, ci și retenția intronului și alte evenimente de îmbinare.
Exemple
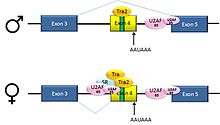
Exon sărind: Drosophila dsx
Pre-ARNm din gena D. melanogaster dsx conțin 6 exoni. La bărbați, exonii 1,2,3,5 și 6 sunt uniți pentru a forma ARNm, care codifică o proteină de reglare transcripțională necesară pentru dezvoltarea masculină. La femele, exonii 1,2,3 și 4 sunt uniți, iar un semnal de poliadenilare în exonul 4 determină scindarea ARNm în acel punct. ARNm rezultat este o proteină de reglare transcripțională necesară dezvoltării feminine.
Acesta este un exemplu de exon saltare. Intronul din amonte de la exonul 4 are un tract polipirimidinic care nu se potrivește bine cu secvența consens , astfel încât proteinele U2AF se leagă slab de acesta fără asistența activatorilor de îmbinare. Prin urmare, acest site acceptor de îmbinare de 3 'nu este utilizat la bărbați. Femelele, cu toate acestea, produc activatorul de îmbinare Transformator (Tra) (vezi mai jos). Proteina SR Tra2 este produsă la ambele sexe și se leagă de ESE în exonul 4; dacă Tra este prezent, se leagă de Tra2 și, împreună cu o altă proteină SR, formează un complex care ajută proteinele U2AF să se lege la tractul slab de polipirimidină. U2 este recrutat la punctul ramificat asociat, iar acest lucru duce la includerea exonului 4 în ARNm.
Site-uri de acceptare alternative: Drosophila Transformator
Pre-ARNm-urile genei Transformator (Tra) ale Drosophila melanogaster suferă splicing alternativ prin modul de acceptare alternativă. Gena Tra codifică o proteină care este exprimată numai la femele. Transcriptul primar al acestei gene conține un intron cu două site-uri acceptabile posibile. La bărbați, este utilizat site-ul acceptor în amonte. Aceasta face ca o versiune mai lungă a exonului 2 să fie inclusă în transcrierea procesată, inclusiv un codon de oprire timpurie . ARNm rezultat codifică un produs proteic trunchiat care este inactiv. Femelele produc proteina principală pentru determinarea sexului Sex letal (Sxl). Proteina Sxl este un represor de îmbinare care se leagă de un ISS în ARN-ul transcriptului Tra în apropierea sitului acceptor din amonte, împiedicând proteina U2AF să se lege de tractul polipirimidinic. Acest lucru împiedică utilizarea acestei joncțiuni, mutând legarea spliceozomului la situl acceptor din aval. Împletirea în acest moment ocolește codonul stop, care este excizat ca parte a intronului. ARNm rezultat codifică o proteină Tra activă, care în sine este un regulator al îmbinării alternative a altor gene legate de sex (a se vedea dsx mai sus).
Definiția exonului: receptor Fas
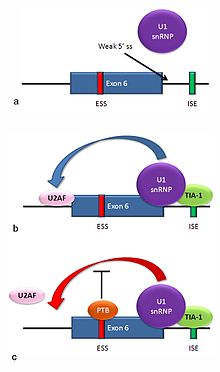
Mai multe izoforme ale proteinei receptorului Fas sunt produse prin îmbinare alternativă. Două izoforme care apar în mod normal la oameni sunt produse de un mecanism de exon-jumping. Un ARNm care include exonul 6 codifică forma legată de membrană a receptorului Fas, care promovează apoptoza sau moartea celulară programată. Expresia crescută a receptorului Fas în celulele pielii expuse cronic la soare și absența expresiei în celulele canceroase ale pielii sugerează că acest mecanism poate fi important în eliminarea celulelor precanceroase la om. Dacă se omite exonul 6, ARNm rezultat codifică o proteină Fas solubilă care nu promovează apoptoza. Includerea sau omiterea exonului depinde de două proteine antagoniste, TIA-1 și proteina de legare a tractului polipirimidinic (PTB).
- Site-ul donator 5 'din intron în aval de exonul 6 din pre-ARNm are un acord slab cu secvența consens și nu este legat de obicei de sn1NR U1. Dacă U1 nu se leagă, exonul este omis (a se vedea „a” în figura însoțitoare).
- Legarea proteinei TIA-1 la un situs de amplificare intronic stabilizează legarea snRNP U1. Complexul rezultat al sitului donator de 5 'ajută la legarea factorului de îmbinare U2AF la locul de îmbinare 3' din amonte de exon, printr-un mecanism care nu este încă cunoscut (vezi b).
- Exonul 6 conține un amortizor de explicare exonic bogat în pirimidină, ure6 , unde PTB se poate lega. Dacă PTB se leagă, acesta inhibă efectul complexului donator 5 'asupra legării U2AF de situsul acceptor, rezultând saltarea exonului (vezi c).
Acest mecanism este un exemplu de definiție a exonului în splicing. Un spliceozom se asamblează pe un intron, iar subunitățile snRNP pliază ARN-ul astfel încât capetele 5 'și 3' ale intronului să fie unite. Cu toate acestea, exemple studiate recent, precum acesta, arată că există și interacțiuni între capetele exonului. În acest caz particular, aceste interacțiuni de definire a exonului sunt necesare pentru a permite legarea factorilor de îmbinare a nucleului înainte de asamblarea spliceozomilor pe cei doi introni flancatori.
Competiție activator represor: HIV-1 tat exon 2
HIV , retrovirusul care cauzează SIDA la om, produce un singur transcript primar de ARN, care este alternativ îmbinat în mai multe moduri pentru a produce peste 40 de mARN diferiți. Echilibrul dintre transcrierile diferențial îmbinate oferă mai mulți ARNm care codifică diferite produse care sunt necesare pentru multiplicarea virală. Unul dintre transcrierile îmbinate diferențial conține gena tat , în care exonul 2 este un exon de casetă care poate fi omis sau inclus. Includerea exonului 2 în ARN este reglementată de concurența dintre represorul de îmbinare hnRNP A1 și proteina SR SC35. În cadrul exonului 2 se suprapun o secvență exonerantă de amortizare a silențierului (ESS) și o secvență de amplificare exonică (ESE). Dacă proteina represor A1 se leagă de ESS, aceasta inițiază legarea cooperativă a mai multor molecule A1, extinzându-se în situsul donor 5 'în amonte de exonul 2 și împiedicând legarea factorului de îmbinare central U2AF35 la tractul polipirimidinic. Dacă SC35 se leagă de ESE, acesta previne legarea A1 și menține site-ul donator 5 'într-o stare accesibilă pentru asamblarea spliceozomului. Concurența dintre activator și represor asigură producerea ambelor tipuri de ARNm (cu și fără exonul 2).
Semnificație adaptativă
Splicarea alternativă este una dintre mai multe excepții de la ideea inițială conform căreia o secvență ADN codifică o polipeptidă ( ipoteza unei gene-o enzimă ). Ar putea fi mai corect acum să spunem „O genă - multe polipeptide”. Sunt necesare informații externe pentru a decide ce polipeptidă este produsă, având în vedere o secvență ADN și pre-ARNm. Deoarece metodele de reglare sunt moștenite, aceasta oferă noi modalități prin care mutațiile pot afecta expresia genelor.
S-a propus că pentru eucariote îmbinarea alternativă a fost un pas foarte important către o eficiență mai mare, deoarece informațiile pot fi stocate mult mai economic. Mai multe proteine pot fi codificate de o singură genă, mai degrabă decât necesitând o genă separată pentru fiecare, permițând astfel un proteom mai variat dintr-un genom de dimensiuni limitate. De asemenea, oferă flexibilitate evolutivă. O mutație cu un singur punct poate determina ocazional un excon dat sau inclus dintr-o transcriere în timpul îmbinării, permițând producerea unei noi izoforme proteice fără pierderea proteinei originale. Studiile au identificat regiuni intrinsec dezordonate (vezi Proteine intrinsec nestructurate ) ca fiind îmbogățite în exonii neconstituționali, sugerând că izoformele proteice pot prezenta diversitate funcțională datorită modificării modulelor funcționale din aceste regiuni. O astfel de diversitate funcțională realizată de izoforme este reflectată de tiparele lor de expresie și poate fi prezisă prin abordări de învățare automată. Studiile comparative indică faptul că îmbinarea alternativă a precedat multicelularitatea în evoluție și sugerează că acest mecanism ar fi putut fi cooptat pentru a ajuta la dezvoltarea organismelor multicelulare.
Cercetările bazate pe Proiectul genomului uman și alte secvențieri ale genomului au arătat că oamenii au doar aproximativ 30% mai multe gene decât viermele rotund Caenorhabditis elegans și doar de aproximativ două ori mai multe decât muștele Drosophila melanogaster . Această constatare a condus la speculații că complexitatea percepută mai mare a oamenilor sau a vertebratelor, în general, s-ar putea datora unor rate mai mari de îmbinare alternativă la om decât la nevertebrate. Cu toate acestea, un studiu pe eșantioane de 100.000 EST, fiecare de la om, șoarece, șobolan, vacă, muscă ( D. melanogaster ), vierme ( C. elegans ) și planta Arabidopsis thaliana nu a găsit diferențe mari în frecvența genelor alternativ îmbinate între oameni și oricare dintre celelalte animale testate. Un alt studiu, cu toate acestea, a propus că aceste rezultate au fost un artefact al diferitelor numere de EST disponibile pentru diferitele organisme. Când au comparat frecvențe de îmbinare alternative în subseturi aleatorii de gene din fiecare organism, autorii au ajuns la concluzia că vertebratele au rate mai mari de îmbinare alternativă decât nevertebratele.
Boală
Modificări ale mașinilor de procesare a ARN pot duce la splicarea greșită a mai multor transcripții, în timp ce modificările cu un singur nucleotid în siturile de îmbinare sau în siturile de reglare cu acționare cis pot duce la diferențe în îmbinarea unei singure gene și, astfel, în ARNm produs dintr-o transcrierile genei mutante. Un studiu din 2005 care a implicat analize probabilistice a indicat că mai mult de 60% dintre mutațiile cauzatoare de boli umane afectează splicing, mai degrabă decât afectează direct secvențele de codificare. Un studiu mai recent indică faptul că o treime din toate bolile ereditare vor avea probabil o componentă de îmbinare. Indiferent de procentul exact, există o serie de boli legate de splicing. Așa cum este descris mai jos, un exemplu proeminent de boli legate de splicing este cancerul.
MARN-urile anormale splicate se găsesc, de asemenea, într-o proporție mare de celule canceroase. Analizele combinate de ARN-Seq și proteomică au dezvăluit o expresie diferențială izbitoare a izoformelor de îmbinare a proteinelor cheie în căile importante de cancer. Nu este întotdeauna clar dacă astfel de modele aberante de îmbinare contribuie la creșterea canceroasă sau sunt doar consecința anomaliilor celulare asociate cu cancerul. Pentru anumite tipuri de cancer, cum ar fi în cazul colorectalului și prostatei, numărul erorilor de îmbinare pe cancer s-a dovedit a varia foarte mult între cancerele individuale, fenomen denumit instabilitate transcriptomică . S-a demonstrat în continuare că instabilitatea transcriptomului corelează grealitatea cu nivelul de expresie redus al genelor factorului de îmbinare. Mutația DNMT3A a fost demonstrat de a contribui la afectiuni maligne hematologice , și că DNMT3A -mutated linii celulare prezintă transcriptome instabilitate în comparație cu omologii lor de tip sălbatic izogene.
De fapt, există de fapt o reducere a îmbinării alternative în celulele canceroase în comparație cu cele normale, iar tipurile de îmbinare diferă; de exemplu, celulele canceroase prezintă niveluri mai ridicate de retenție a intronului decât celulele normale, dar niveluri mai scăzute de sărituri ale exonului. Unele dintre diferențele în splicing în celulele canceroase se pot datora frecvenței ridicate a mutațiilor somatice în genele factorului de splicing, iar unele pot rezulta din modificări în fosforilarea factorilor de splicing cu acțiune trans. Altele pot fi produse prin modificări ale cantităților relative de factori de îmbinare produse; de exemplu, s-a demonstrat că celulele cancerului de sân au niveluri crescute ale factorului de îmbinare SF2 / ASF . Un studiu a constatat că un procent relativ mic (383 din peste 26000) de variante alternative de splicing au fost semnificativ mai mari în frecvență în celulele tumorale decât celulele normale, sugerând că există un set limitat de gene care, atunci când sunt splicate greșit, contribuie la apariția tumorii dezvoltare. Cu toate acestea, se crede că efectele dăunătoare ale transcrierilor greșite sunt de obicei protejate și eliminate printr-un mecanism de control al calității posttranscripțional celular denumit dezintegrare a ARNm mediată prin prostii [NMD].
Un exemplu de variantă specifică de îmbinare asociată cu cancerele se află într-una dintre genele DNMT umane . Trei gene DNMT codifică enzime care adaugă grupări metil la ADN, o modificare care are adesea efecte de reglare. Mai multe mARN-uri DNMT3B anexate anormal se găsesc în tumori și în liniile celulare canceroase. În două studii separate, exprimarea a două dintre aceste mARN-uri anormale îmbinate în celulele de mamifere au provocat modificări ale modelelor de metilare a ADN-ului în acele celule. Celulele cu unul dintre mARN-urile anormale au crescut, de asemenea, de două ori mai repede decât celulele martor, indicând o contribuție directă la dezvoltarea tumorii de către acest produs.
Un alt exemplu este proto- oncogenul Ron ( MST1R ) . O proprietate importantă a celulelor canceroase este capacitatea lor de a se mișca și de a invada țesutul normal. S-a constatat că producția unui transcript anormal de splicat al lui Ron este asociată cu niveluri crescute de SF2 / ASF în celulele cancerului de sân. Izoforma anormală a proteinei Ron codificată de acest ARNm duce la motilitatea celulară .
Supraexprimarea unei variante splice trunchiată a FOSB genei - ΔFosB - într - o anumită populație de neuroni din nucleul accumbens a fost identificat ca mecanismul cauzal implicat în inducerea și menținerea unei dependenta de droguri si recompense naturale .
Studii recente provocatoare indică o funcție cheie a structurii cromatinei și a modificărilor histonice în reglarea alternativă a îmbinării. Aceste perspective sugerează că reglarea epigenetică determină nu numai ce părți ale genomului sunt exprimate, ci și modul în care sunt îmbinate.
Analiza la nivel de genom
Analiza la nivel de genom a îmbinării alternative este o sarcină provocatoare. De obicei, au fost găsite transcripții combinate alternativ prin compararea secvențelor EST , dar acest lucru necesită secvențierea unui număr foarte mare de EST. Majoritatea bibliotecilor EST provin dintr-un număr foarte limitat de țesuturi, astfel încât este posibil ca variantele de îmbinare specifice țesutului să fie omise în orice caz. Au fost, totuși, dezvoltate abordări de mare viteză pentru investigarea splicingului, cum ar fi: analize bazate pe microarray de ADN , teste de legare a ARN și secvențierea profundă . Aceste metode pot fi utilizate pentru a depista polimorfismele sau mutațiile în sau în jurul elementelor de îmbinare care afectează legarea proteinelor. Atunci când sunt combinate cu teste de splicing, inclusiv teste de gene reporter in vivo , efectele funcționale ale polimorfismelor sau mutațiilor asupra splicingului transcrierilor pre-ARNm pot fi apoi analizate.
În analiza microarray, au fost utilizate matrici de fragmente de ADN care reprezintă exoni individuali (de exemplu, microarray exon Affymetrix ) sau limite exon / exon (de exemplu matrici de la ExonHit sau Jivan ). Matricea este apoi testată cu ADNc marcat din țesuturile de interes. ADNc-urile sondei se leagă de ADN-ul de la exonii care sunt incluși în ARNm în țesutul lor de origine sau de ADN-ul de la limita unde au fost uniți doi exoni. Acest lucru poate dezvălui prezența anumitor ARNm spliced alternativ.
CLIP ( Cross-linking and immunoprecipitation ) folosește radiațiile UV pentru a lega proteinele de moleculele de ARN dintr-un țesut în timpul îmbinării. O proteină de reglare cu acțiune trans-acționantă de interes este apoi precipitată folosind anticorpi specifici. Când ARN-ul atașat la proteina respectivă este izolat și clonat, acesta dezvăluie secvențele țintă pentru proteina respectivă. O altă metodă de identificare a proteinelor care leagă ARN și de cartografiere a legării acestora la transcripțiile pre-ARNm este „Evaluarea microarray a Aptamerilor genomici prin schimbare (MEGAshift)”. Net Această metodă implică o adaptare a „Evoluției sistematice a liganzilor prin îmbogățire exponențială (SELEX) "metoda împreună cu o citire bazată pe microarrays. Utilizarea metodei MEGAshift a oferit informații despre reglementarea splicingului alternativ, permițând identificarea secvențelor din transcripțiile pre-ARNm care înconjoară exonii spliced alternativi care mediază legarea la diferiți factori de splicing, cum ar fi ASF / SF2 și PTB. Această abordare a fost, de asemenea, utilizată pentru a ajuta la determinarea relației dintre structura secundară a ARN și legarea factorilor de îmbinare.
Tehnologiile de secvențiere profundă au fost folosite pentru a efectua analize la nivel genomic ale ARNm - neprocesate și procesate - oferind astfel informații despre îmbinarea alternativă. De exemplu, rezultatele utilizării secvențierii profunde indică faptul că, la om, aproximativ 95% din transcrierile din genele multiexon suferă o îmbinare alternativă, cu un număr de transcripții pre-ARNm s-au îmbinat într-o manieră specifică țesutului. Genomica funcțională și abordările de calcul bazate pe învățarea în mai multe instanțe au fost, de asemenea, dezvoltate pentru a integra datele ARN-seq pentru a prezice funcțiile pentru izoforme alternative îmbinate. Secvențierea profundă a ajutat, de asemenea, la detectarea in vivo a lariaturilor tranzitorii care sunt eliberate în timpul splicingului, determinarea secvențelor site-ului ramurilor și cartografierea pe scară largă a punctelor ramificate în transcripțiile pre-ARNm umane.
Utilizarea analizelor de reporter face posibilă găsirea proteinelor de splicing implicate într-un eveniment specific de splicing alternativ prin construirea de gene reporter care vor exprima una din cele două proteine fluorescente diferite în funcție de reacția de splicing care are loc. Această metodă a fost utilizată pentru a izola mutanții care afectează splicing-ul și astfel pentru a identifica proteine reglatoare noi de splicing inactivate în acei mutanți.
Baze de date
Există o colecție de baze de date de îmbinare alternative. Aceste baze de date sunt utile pentru găsirea genelor care au pre-ARNm care suferă splicing alternativ și evenimente de splicing alternativ sau pentru a studia impactul funcțional al splicing alternativ.
Vezi si
Referințe
linkuri externe
- O definiție generală și o nomenclatură pentru evenimente alternative de îmbinare la SciVee
- AStalavista (Instrument de vizualizare peisaj alternativ Splicing), o metodă pentru clasificarea computerizată exhaustivă a structurilor alternative Splicing
- IsoPred: funcții isoforme prezise prin calcul
- Stamms-lab.net: Grup de cercetare care se ocupă de problemele alternative de splicing și de splicing greșit în bolile umane
- Alternativă Splicing de canale ionice în creier, conectate la boli mentale și neurologice
- BIPASS: Servicii Web în Splicing alternativ